Código
# install
install.packages("ggplot2")
# load library
library(ggplot2)5 de noviembre de 2024
Comprender la lógica detras de la estructura los gráficos con ggplot2
Ser capaz de intregrar en un gŕafico diferentes fuentes de información que representen la complejidad de los datos
Un paquete de R diseñado específicamente para producir gráficos
A diferencia de otros paquetes, ggplot2 tiene su propia gramática
La gramática se basa en la “Gramática de los gráficos” (Wilkinson 2005)
Módulos independientes que pueden combinarse de muchas formas
Esta gramática proporciona una gran flexibilidad
La idea principal es empezar con una capa base de datos brutos y luego añadir más capas de anotaciones y resúmenes estadísticos. El paquete nos permite producir gráficos utilizando la misma estructura de pensamiento que utilizamos al diseñar un análisis, reduciendo la distancia de cómo visualizamos un gráfico en la cabeza y el producto final.
Aprender la gramática no sólo es útil para producir un gráfico de interés, sino también para pensar en otros gráficos más complejos. La ventaja de esta gramática es la posibilidad de crear nuevos gráficos compuestos por nuevas combinaciones de elementos.
Todos los gráficos ggplot2 contienen los siguientes componentes:
Estos componentes se juntan utilizando “+”.
La sintaxis más habitual incluye los datos dentro de la llamada “ggplot” y una capa “geom_”.
Primero instala/carga el paquete:
Utilicemos el conjunto de datos “iris” para crear gráficos de dispersión:
This plot is defined by 3 components: 1. “data”- iris 1. “aes” - Sepal.length vs Petal.length 1. “layer” - Points (geom)
También podemos añadir otros atributos estéticos como el color, la forma y el tamaño. Estos atributos se pueden incluir dentro de aes():
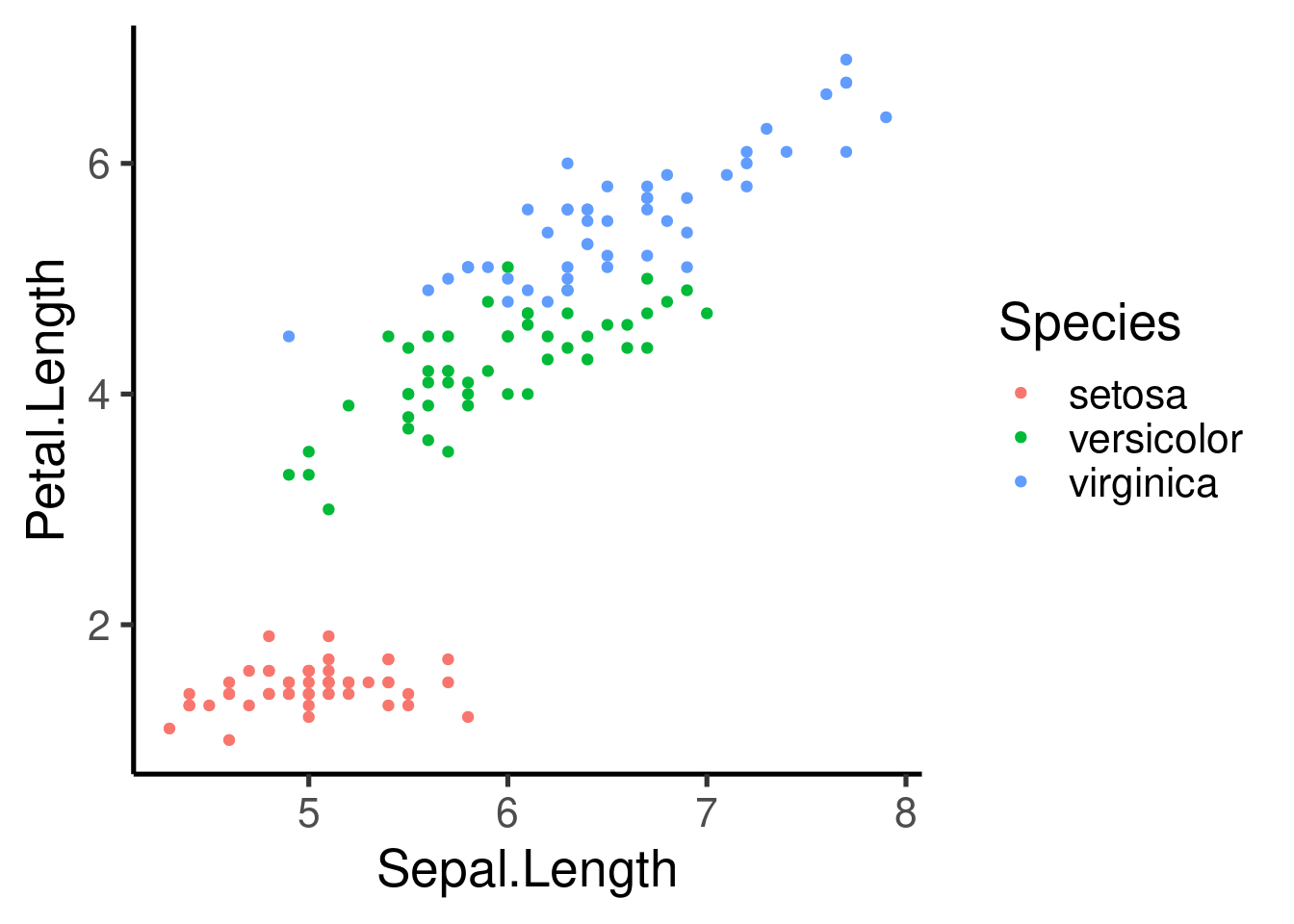
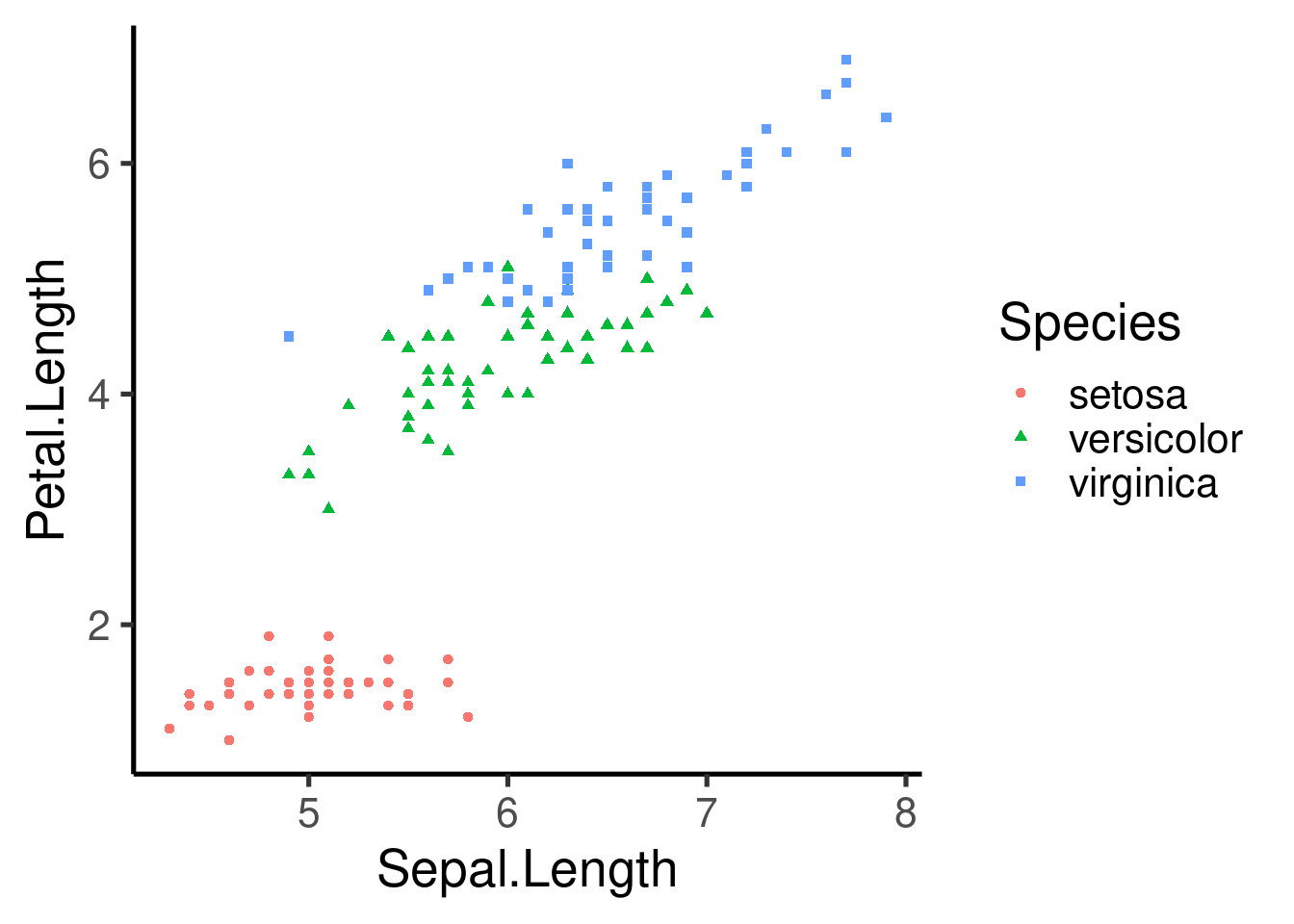
Tenga en cuenta que los argumentos estéticos también pueden incluirse en la capa “geom”:
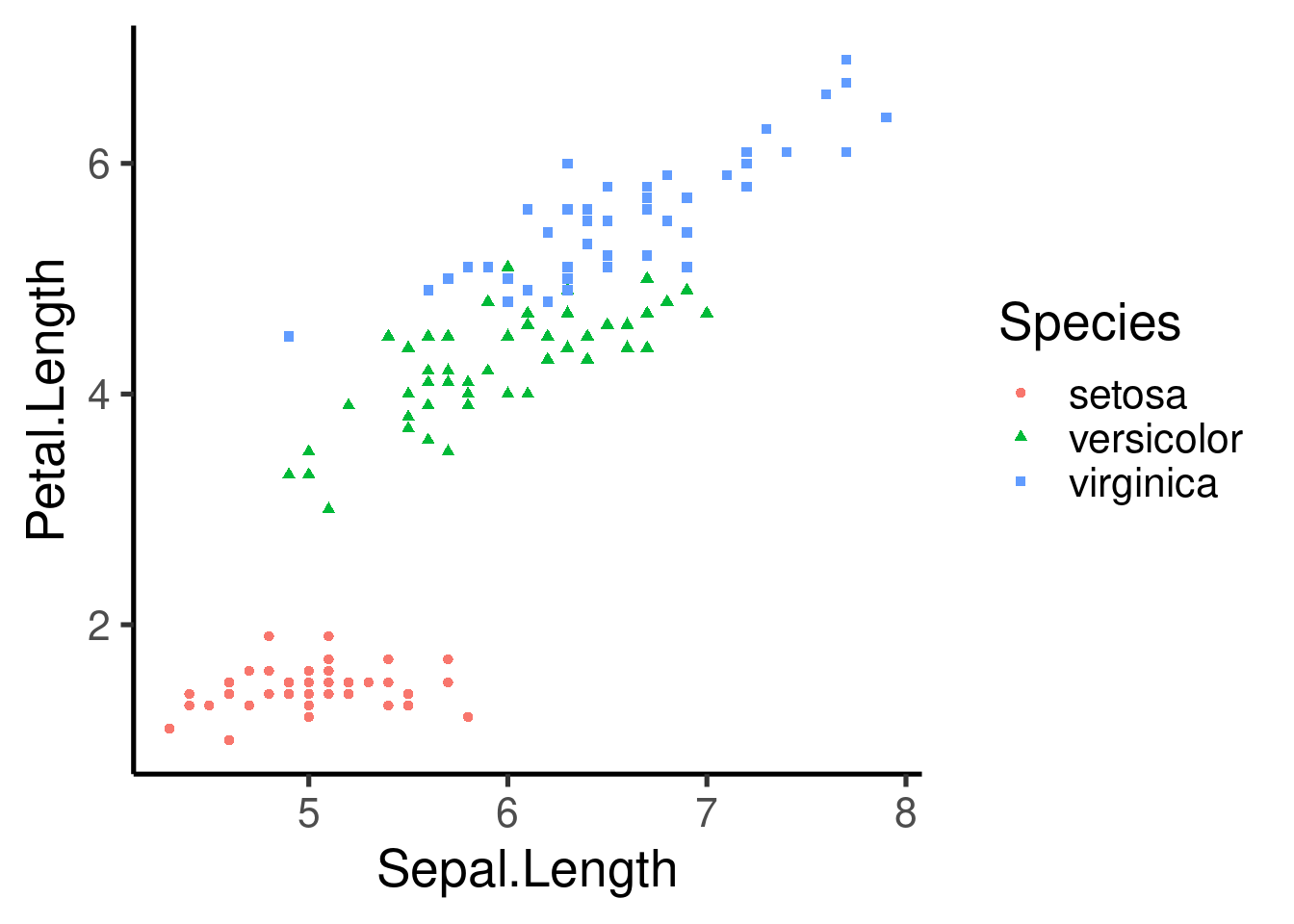
También podemos incluir un valor fijo:
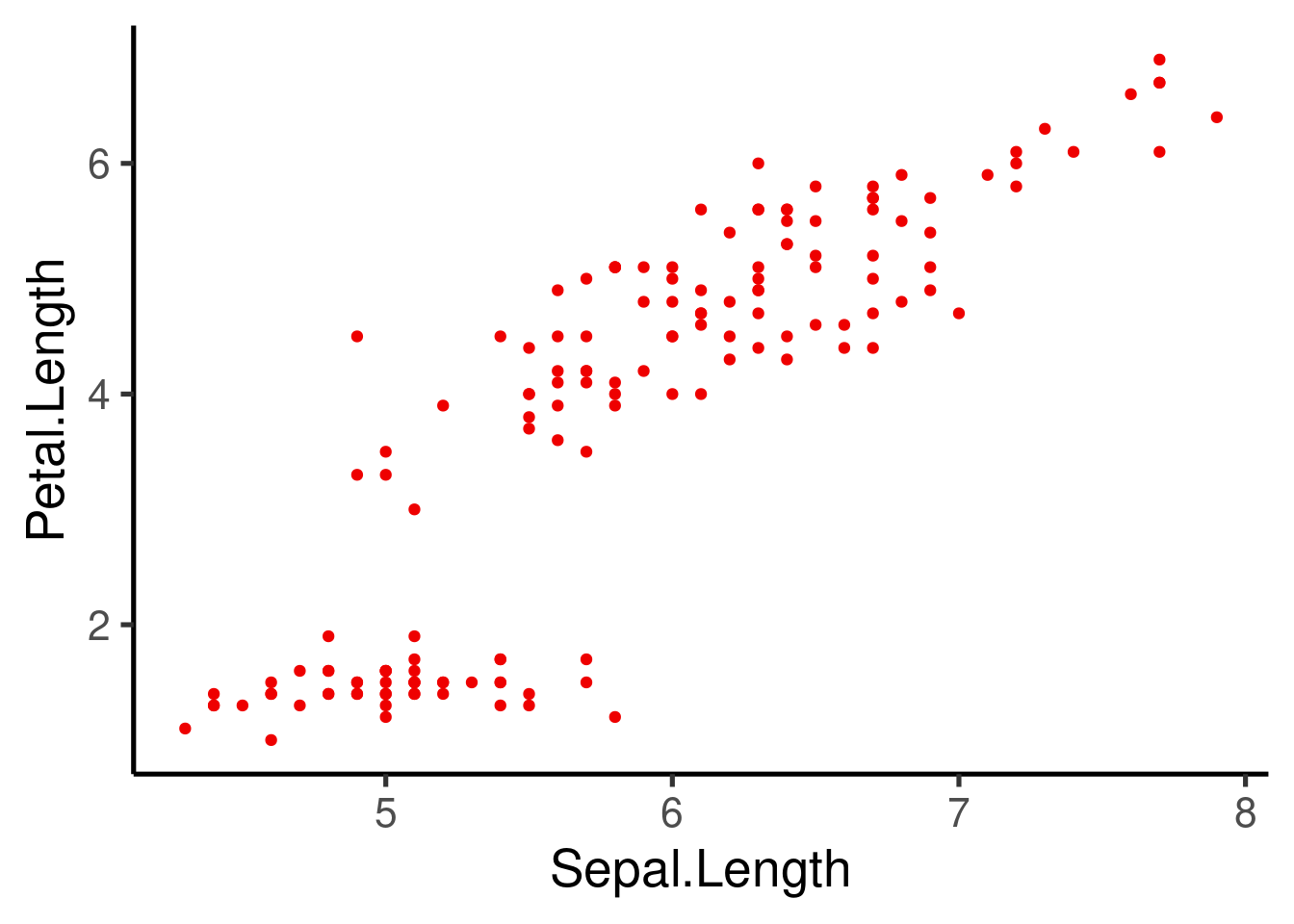
Algunos atributos funcionan mejor con algunos tipos de datos:
Ejercicio 1
Utilizando el conjunto de datos “hylaeformis”:
# lear desde el sitio del curso
hylaeformis_data <- read.csv(
paste0("https://raw.githubusercontent.com/maRce10/", "r_avanzado_2023/master/data/hylaeformis_data.csv")
)
# or bajar manualmente y leer copia local
hylaeformis_data <- read.csv("hylaeformis_data.csv", stringsAsFactors = FALSE)
head(hylaeformis_data, 20)1.1 Create a scatter plot of vs “meanfreq” (mean frequency)
1.1 Crear un gráfico de dispersión de duración (“duration”) vs frecuencia promedio (“meanfreq”)
1.2 Añadir un atributo estético para mostrar un color diferente para cada localidad
1.3 Añade otro atributo estético para mostrar el rango de frecuencia dominante (“dfrange”) como tamaño los símbolos
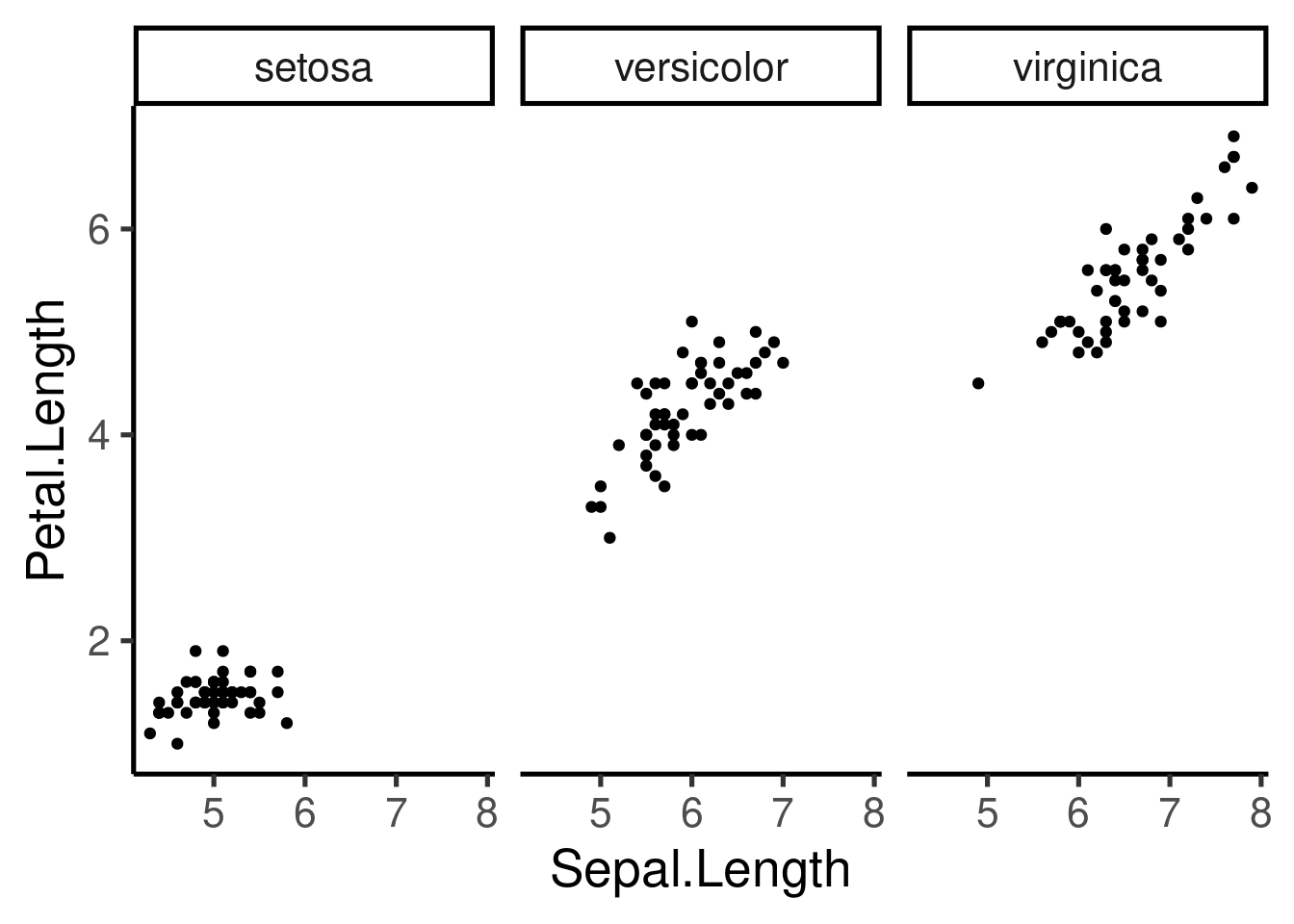
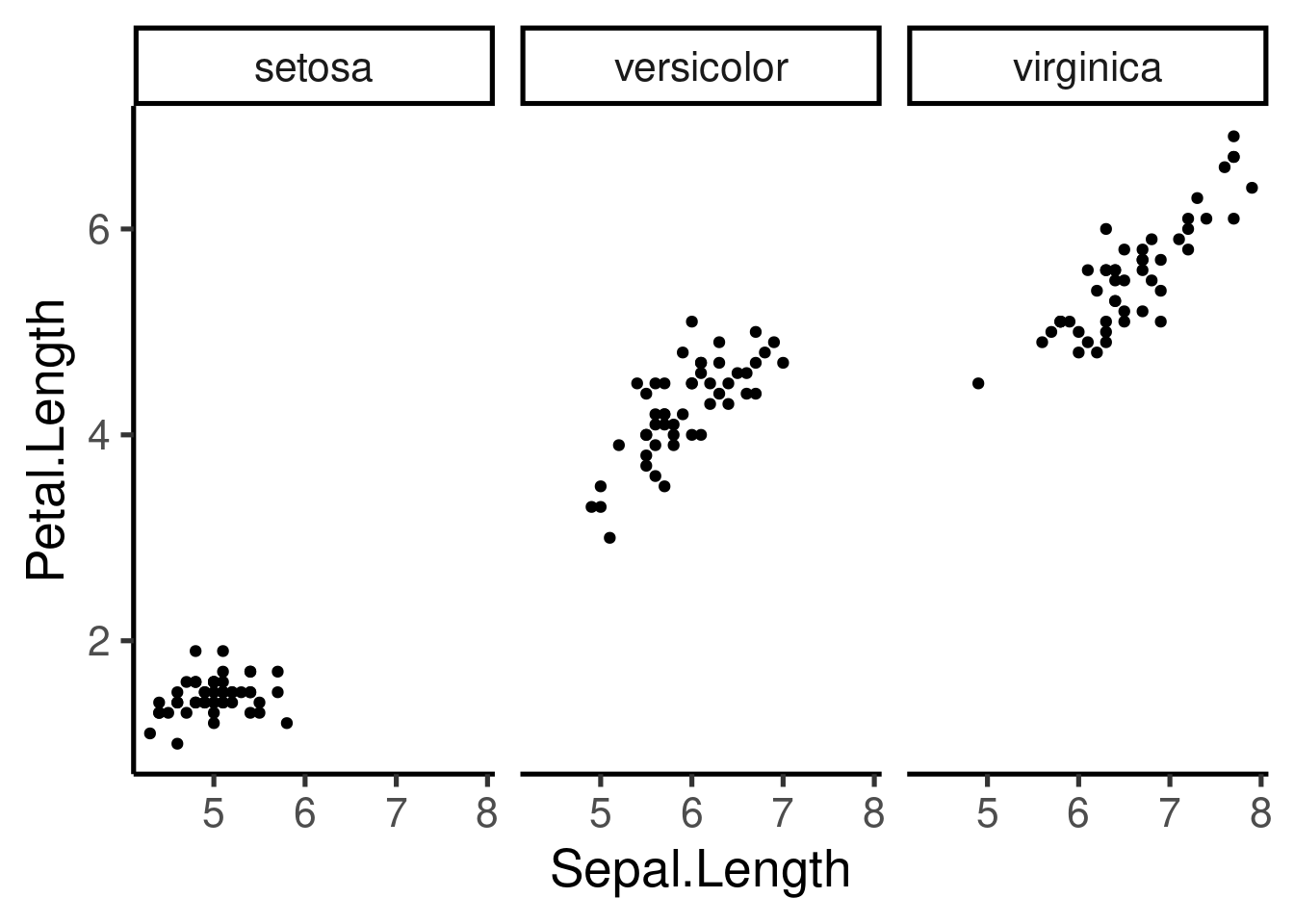
La escala puede ser fija o libre para los ejes x e y, y puede modificarse el número de columnas y filas:
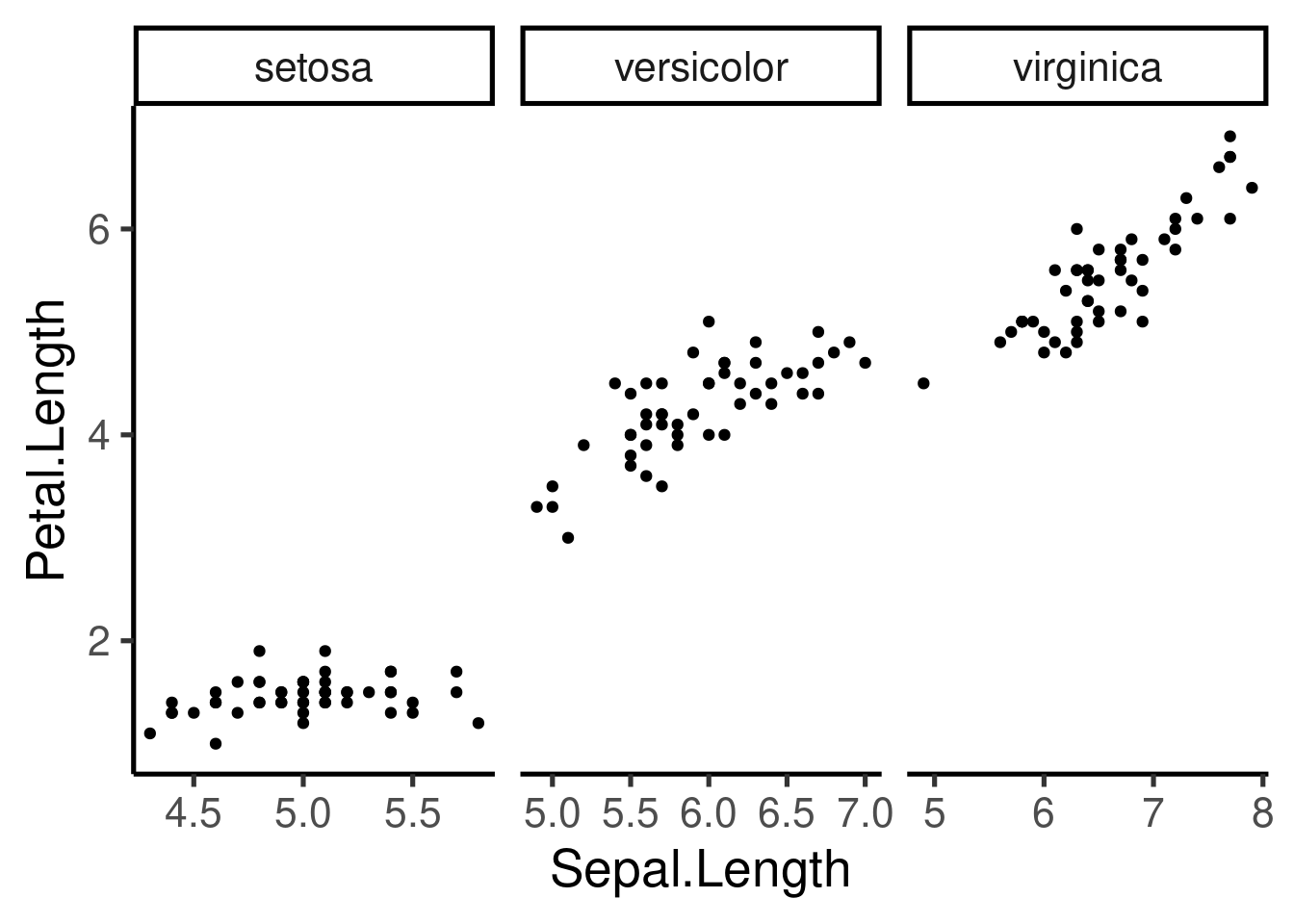
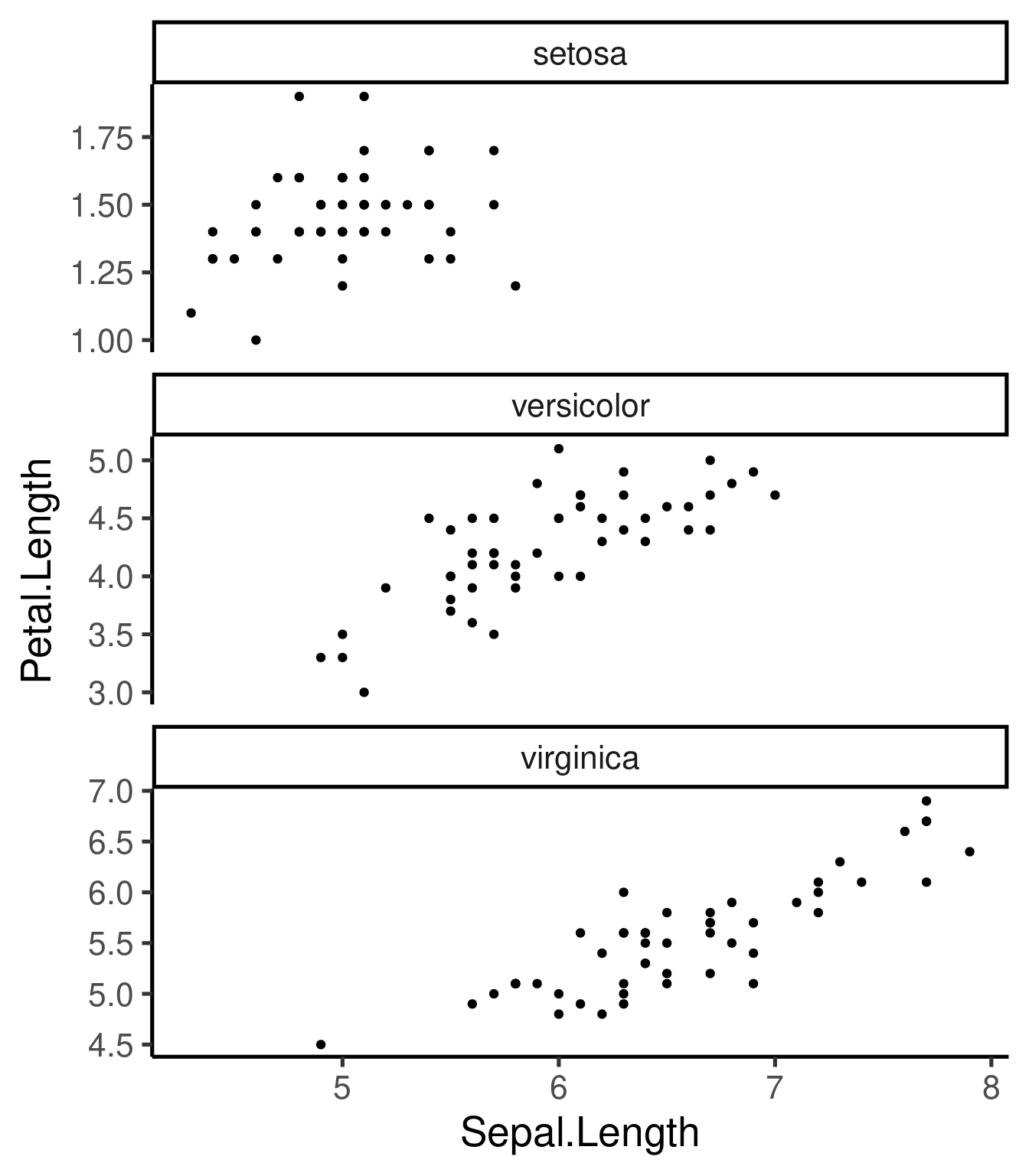
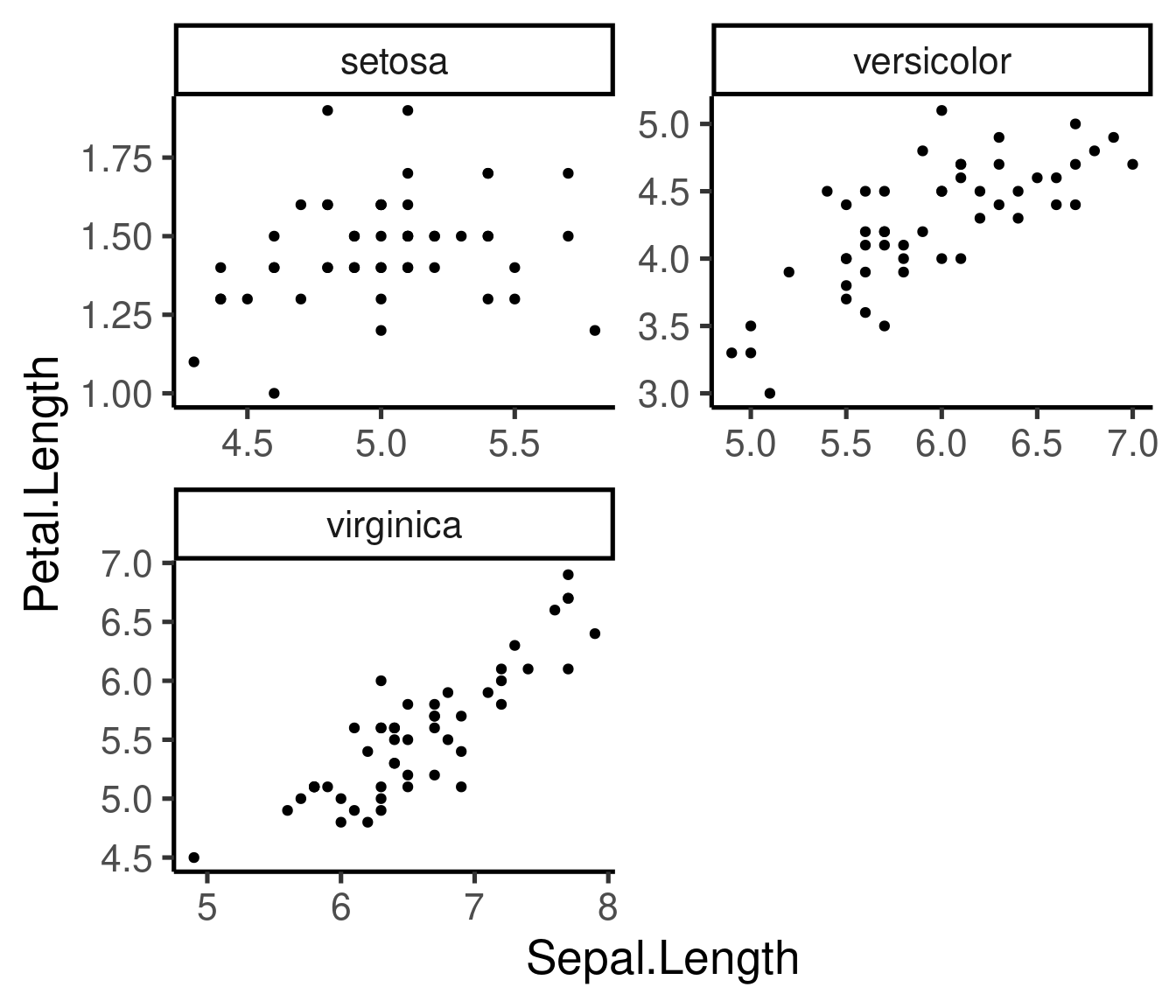
Tenga en cuenta que también podemos guardar el componente básico como un objeto R y añadir otros componentes más adelante en el código:
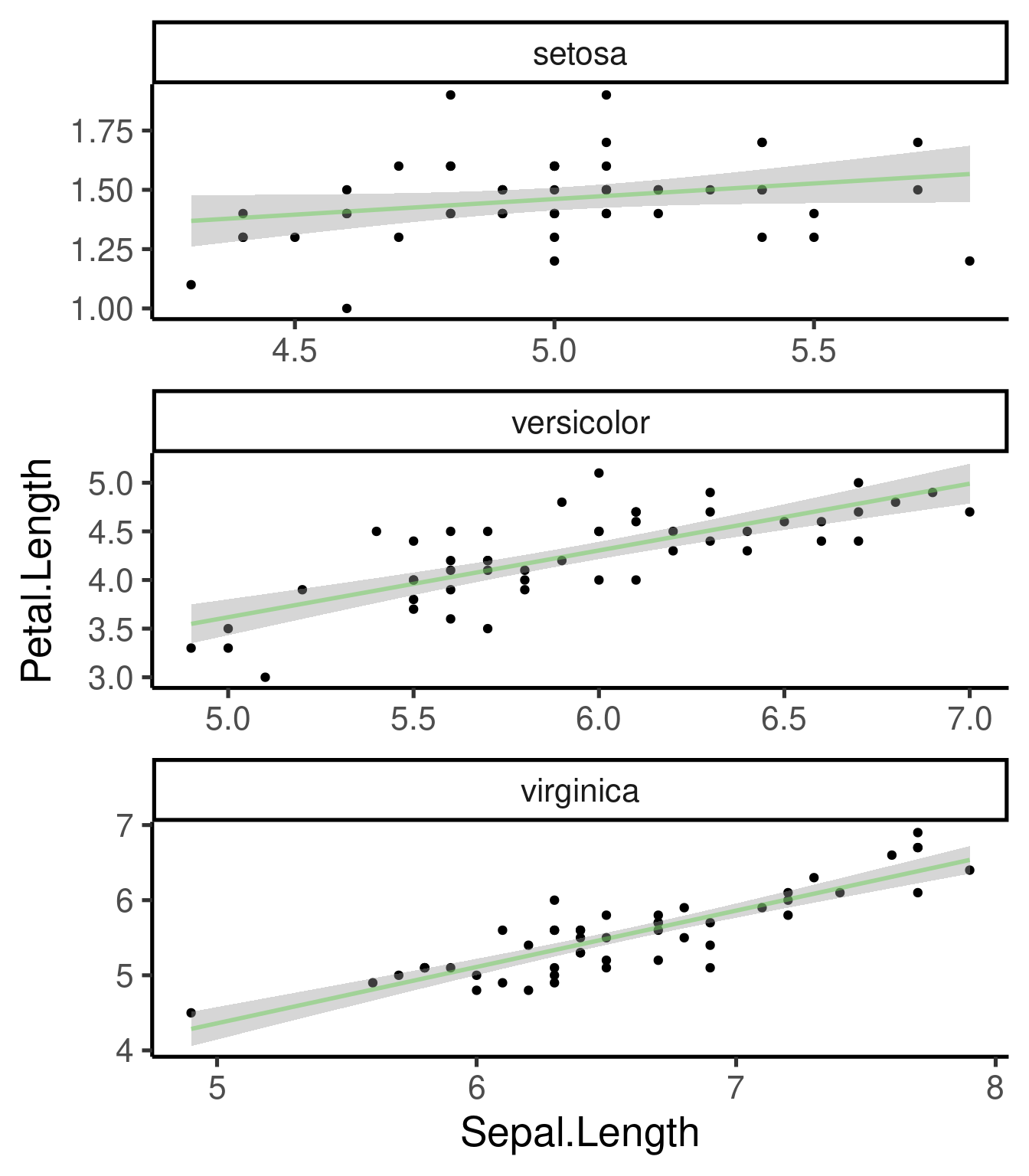
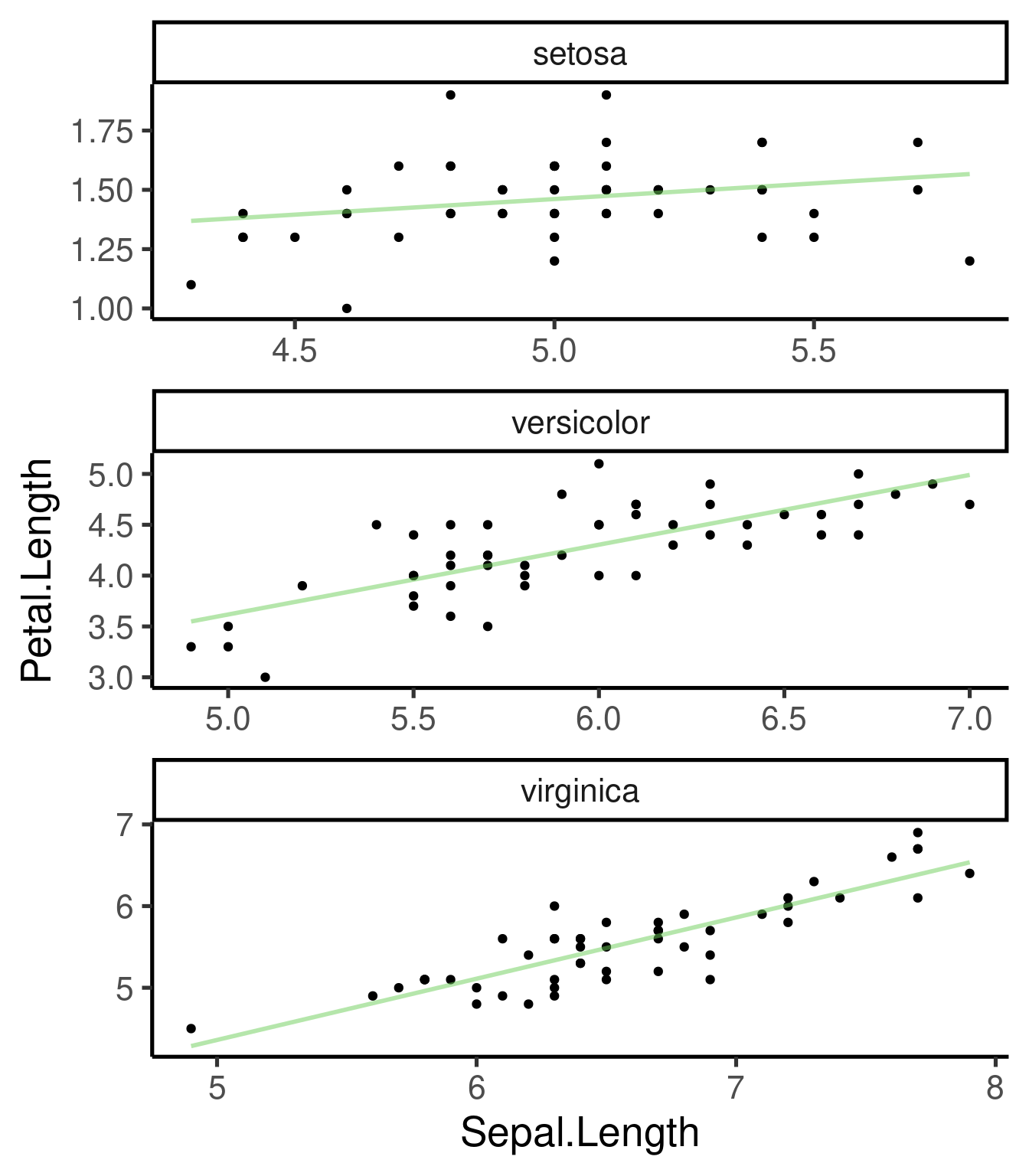
geom_smooth() - añade las líneas de mejor ajuste (incluyendo CI)geom_boxplot() - Distribución de frecuenciasgeom_histogram() & geom_freqpoly() - distribuciones de frecuenciageom_bar() - distribución de frecuencias de variables categóricasgeom_path() & geom_line() - añade líneas a los gráficos de dispersión
Las líneas de regresión de mejor ajuste pueden añadirse con geom_smooth():
Ejercicio 2
Utilizando el conjunto de datos de ejemplo “msleep”:
2.1 Crear un gráfico de dispersión de peso corporal (“bodywt”) frente a peso cerebral (“brainwt”)
2.2 Añadir orden (“order”) como estética del color
2.3 Añadir un componente “faceta” para dividir los gráficos por orden utilizando escalas libres
2.4 Elimine los órdenes con menos de 4 especies en el conjunto de datos y haga un gráfico similar al 2.3
2.5 Añadir una línea de mejor ajuste a cada gráfico del panel
De nuevo, sólo se necesita un nuevo componente “geom” para crear un boxplot:
Una alternativa interesante son los gráficos de violines:
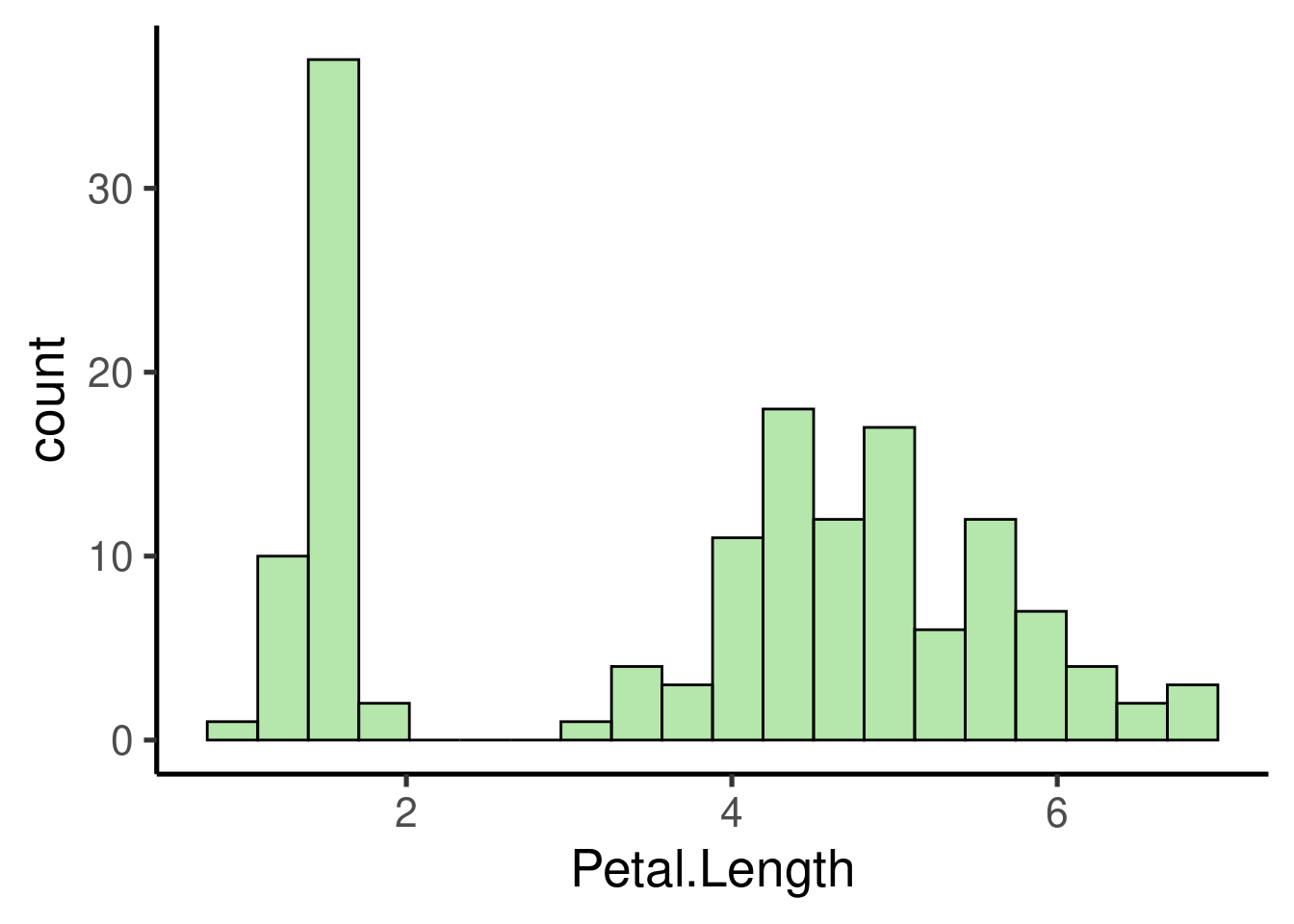
Lo mismo ocurre con los histrogramas y los gráficos de frecuencias:
Podemos controlar la anchura de las barras:
Warning: Duplicated aesthetics after name standardisation: fill
Y compara la distribución de los distintos grupos dentro del mismo histograma:
Muestran la distribución de variables discretas (categóricas):
Además de las funciones básicas (por ejemplo, componentes) descritas anteriormente, ggplot tiene muchas otras herramientas (tanto argumentos como funciones adicionales) para personalizar aún más los gráficos. Prácticamente todo se puede modificar. Aquí vemos algunas de las herramientas más comunes.
ggplot2 viene con algunos temas por defecto que se pueden aplicar fácilmente para modificar el aspecto de nuestros gráficos:
La mayoría de los temas difieren en el uso de cuadrículas, líneas de borde y patrones de etiquetado de ejes.
Los límites de los ejes pueden modificarse como sigue:
Los ejes también pueden transformarse:
o invertidos:
Los ggplots pueden exportarse como archivos de imagen utilizando la función ggsave:
Warning: Removed 21 rows containing missing values or values outside the scale range
(`geom_point()`).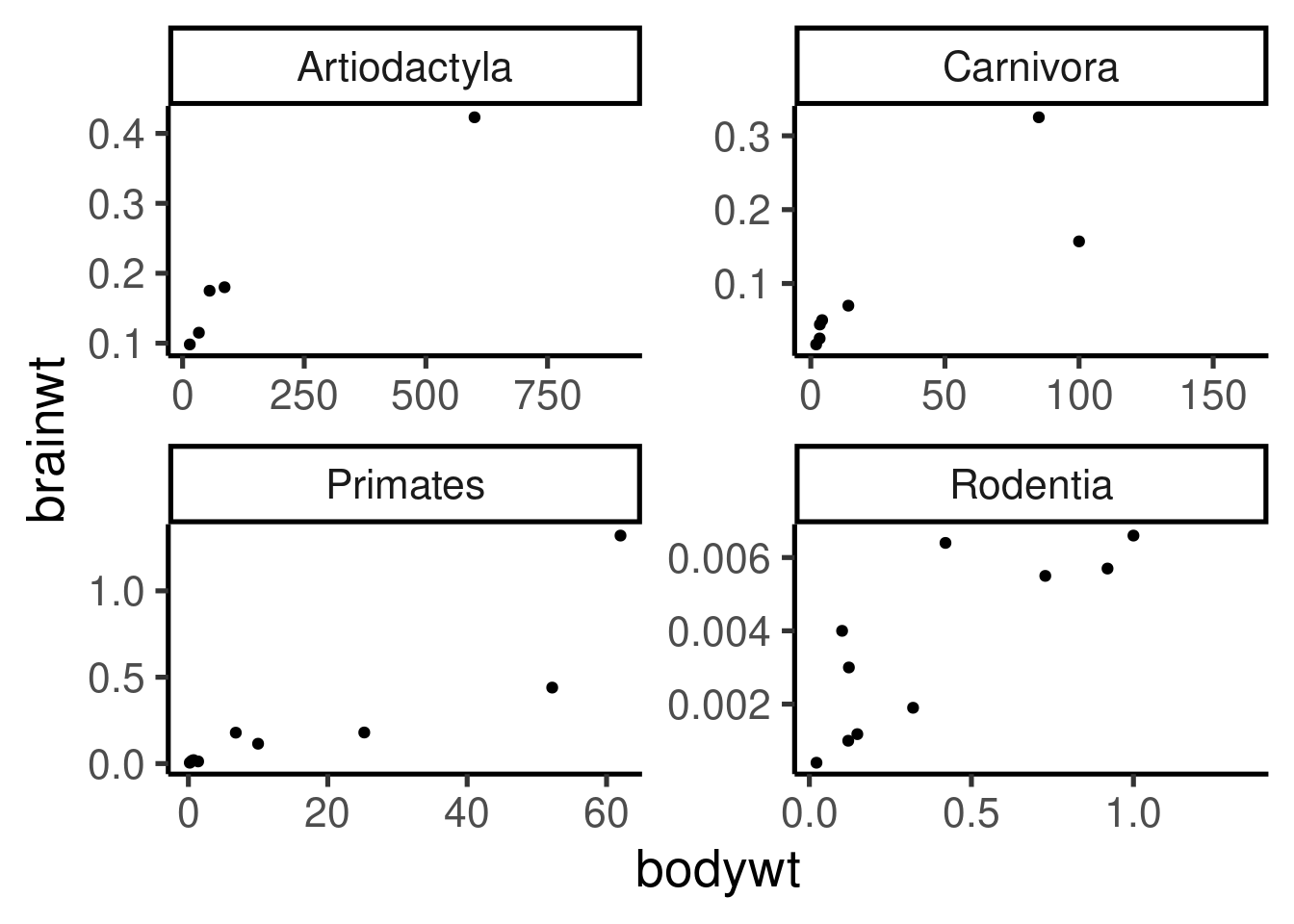
Warning: Removed 21 rows containing missing values or values outside the scale range
(`geom_point()`).
El tipo de archivo de imagen se identificará por la extensión en el nombre del archivo
Personalización adicional del eje:
### Agregar "tick marks" ###
# Cargar librerías
library(MASS)
data(Animals)
# x and y axis are transformed and formatted
p2 <- ggplot(Animals, aes(x = body, y = brain)) +
geom_point(size = 4) +
scale_x_log10(
breaks = trans_breaks("log10", function(x) 10^x),
labels = trans_format("log10", math_format(10^.x))
) +
scale_y_log10(
breaks = trans_breaks("log10", function(x) 10^x),
labels = trans_format("log10", math_format(10^.x))
) +
theme_bw()
# log-log plot without log tick marks
p2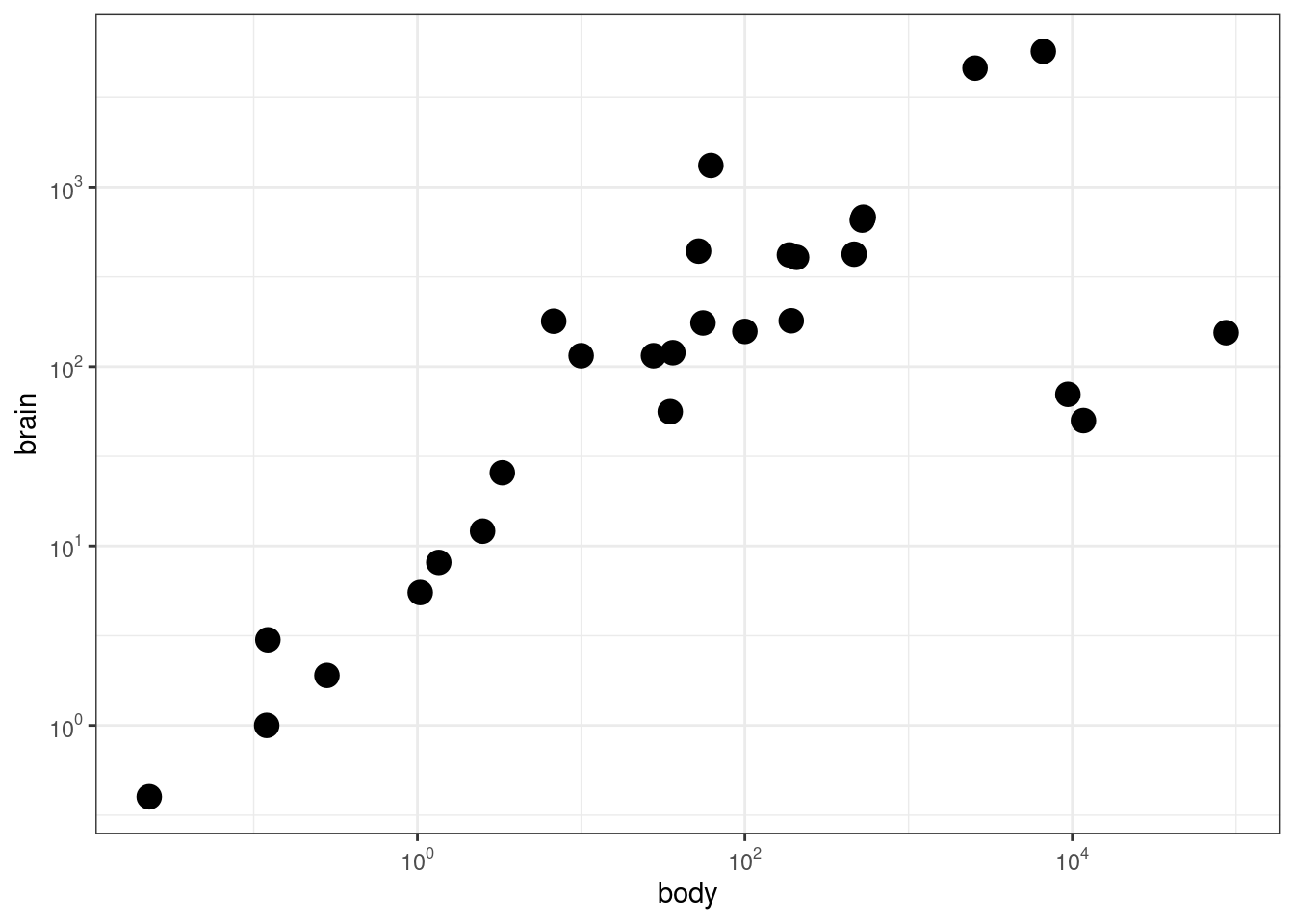
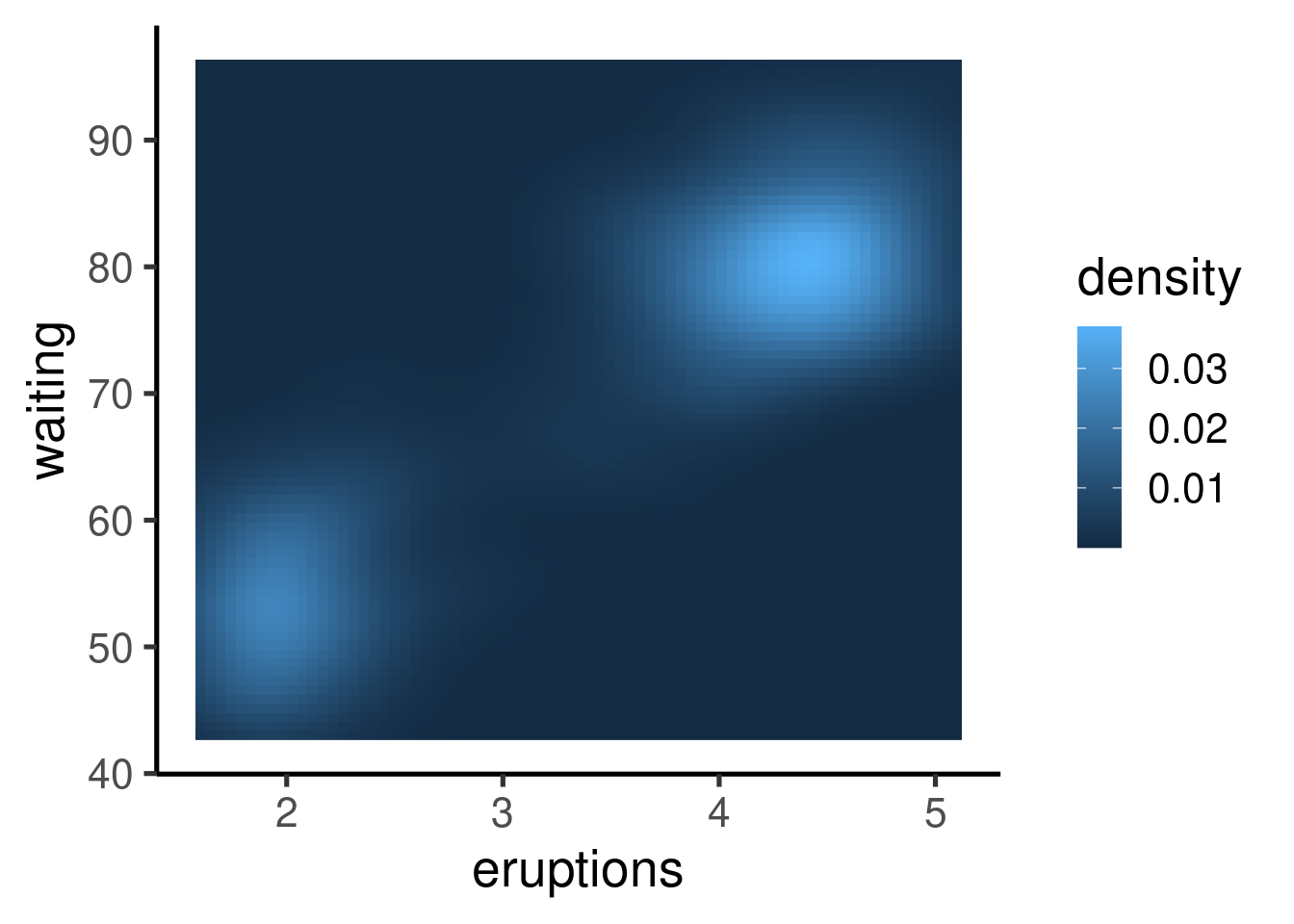
Se pueden generar muchos otros tipos de gráficos. Aquí muestro un ejemplo de gráficos de contorno y de “mapa de calor”:
| eruptions | waiting |
|---|---|
| 3.600 | 79 |
| 1.800 | 54 |
| 3.333 | 74 |
| 2.283 | 62 |
| 4.533 | 85 |
| 2.883 | 55 |
Consulte la CRAN Graphics Task View para obtener una lista más completa de herramientas gráficas en R.
Información de la sesión
R version 4.4.1 (2024-06-14)
Platform: x86_64-pc-linux-gnu
Running under: Ubuntu 22.04.4 LTS
Matrix products: default
BLAS: /usr/lib/x86_64-linux-gnu/blas/libblas.so.3.10.0
LAPACK: /usr/lib/x86_64-linux-gnu/lapack/liblapack.so.3.10.0
locale:
[1] LC_CTYPE=en_US.UTF-8 LC_NUMERIC=C
[3] LC_TIME=es_CR.UTF-8 LC_COLLATE=en_US.UTF-8
[5] LC_MONETARY=es_CR.UTF-8 LC_MESSAGES=en_US.UTF-8
[7] LC_PAPER=es_CR.UTF-8 LC_NAME=C
[9] LC_ADDRESS=C LC_TELEPHONE=C
[11] LC_MEASUREMENT=es_CR.UTF-8 LC_IDENTIFICATION=C
time zone: America/Costa_Rica
tzcode source: system (glibc)
attached base packages:
[1] stats graphics grDevices utils datasets methods base
other attached packages:
[1] scales_1.3.0 viridis_0.6.5 viridisLite_0.4.2 MASS_7.3-61
[5] RColorBrewer_1.1-3 ggplot2_3.5.1 knitr_1.48 kableExtra_1.4.0
loaded via a namespace (and not attached):
[1] Matrix_1.7-0 gtable_0.3.5 jsonlite_1.8.9 dplyr_1.1.4
[5] compiler_4.4.1 tidyselect_1.2.1 xml2_1.3.6 stringr_1.5.1
[9] gridExtra_2.3 textshaping_0.4.0 splines_4.4.1 systemfonts_1.1.0
[13] yaml_2.3.10 fastmap_1.2.0 lattice_0.22-6 R6_2.5.1
[17] labeling_0.4.3 generics_0.1.3 isoband_0.2.7 htmlwidgets_1.6.4
[21] tibble_3.2.1 munsell_0.5.1 svglite_2.1.3 pillar_1.9.0
[25] rlang_1.1.4 utf8_1.2.4 stringi_1.8.4 xfun_0.48
[29] cli_3.6.3 mgcv_1.9-1 withr_3.0.1 magrittr_2.0.3
[33] digest_0.6.37 grid_4.4.1 rstudioapi_0.16.0 nlme_3.1-165
[37] lifecycle_1.0.4 vctrs_0.6.5 evaluate_1.0.1 glue_1.8.0
[41] farver_2.1.2 ragg_1.3.2 fansi_1.0.6 colorspace_2.1-1
[45] rmarkdown_2.28 tools_4.4.1 pkgconfig_2.0.3 htmltools_0.5.8.1---
title: <font size="7"><b>Gráficos con ggplot2</b></font>
---
```{r,echo=FALSE,message=FALSE}
options("digits" = 5)
options("digits.secs" = 3)
# options to customize chunk outputs
knitr::opts_chunk$set(
message = FALSE
)
x <- c("kableExtra", "knitr", "ggplot2", "RColorBrewer", "knitr", "MASS", "viridis")
aa <- lapply(x, function(y) {
if (!y %in% installed.packages()[, "Package"]) {
if (!y %in% c("warbleR", "Rraven")) install.packages(y) else devtools::install_github(paste0("maRce10/", y))
}
try(require(y, character.only = T), silent = T)
})
# ggplot settings
geom_histogram <- function(...) ggplot2::geom_histogram(..., fill = viridis(10, alpha = 0.5)[8], show.legend = FALSE, bins = 20, color = "black")
geom_smooth <- function(...) ggplot2::geom_smooth(..., color = viridis(10, alpha = 0.5)[8])
geom_boxplot <- function(...) ggplot2::geom_boxplot(..., fill = viridis(10, alpha = 0.5)[7])
theme_set(theme_classic(base_size = 20))
```
```{r functions and data, eval=T, echo=F}
hylaeformis_data <- read.csv(
paste0("https://raw.githubusercontent.com/maRce10/", "r_avanzado_2023/master/data/hylaeformis_data.csv")
)
```
::: {.alert .alert-info}
# Objetivos del manual {.unnumbered .unlisted}
- Comprender la lógica detras de la estructura los gráficos con ggplot2
- Ser capaz de intregrar en un gŕafico diferentes fuentes de información que representen la complejidad de los datos
:::
::: {.alert .alert-warning}
## ggplot2 {.unnumbered .unlisted}
- Un paquete de R diseñado específicamente para producir gráficos
- A diferencia de otros paquetes, ggplot2 tiene su propia gramática
- La gramática se basa en la "Gramática de los gráficos" (Wilkinson 2005)
- Módulos independientes que pueden combinarse de muchas formas
- Esta gramática proporciona una gran flexibilidad
:::
## Gramática de gráficos
La idea principal es empezar con una capa base de datos brutos y luego añadir más capas de anotaciones y resúmenes estadísticos. El paquete nos permite producir gráficos utilizando la misma estructura de pensamiento que utilizamos al diseñar un análisis, reduciendo la distancia de cómo visualizamos un gráfico en la cabeza y el producto final.
Aprender la gramática no sólo es útil para producir un gráfico de interés, sino también para pensar en otros gráficos más complejos. La ventaja de esta gramática es la posibilidad de crear nuevos gráficos compuestos por nuevas combinaciones de elementos.
## Componentes del gráfico
Todos los gráficos ggplot2 contienen los siguientes componentes:
- **Datos** - El objeto R con la información que necesita ser trazada
- **Capas** - Los datos específicos que serán graficados (ej. 'x' & 'y')
- **Escala** - Rango de datos a incluir
- **Coordenadas** - Sistema de coordenadas (no se utiliza muy a menudo)
- **Parcelas (facets)** - Determina cómo dividir los datos en subparcelas en un multipanel
- **Tema** - Controla el estilo del gráfico
Estos componentes se juntan utilizando "+".
La sintaxis más habitual incluye los datos dentro de la llamada "ggplot" y una capa "geom\_".
Primero instala/carga el paquete:
```{r, eval=FALSE}
# install
install.packages("ggplot2")
# load library
library(ggplot2)
```
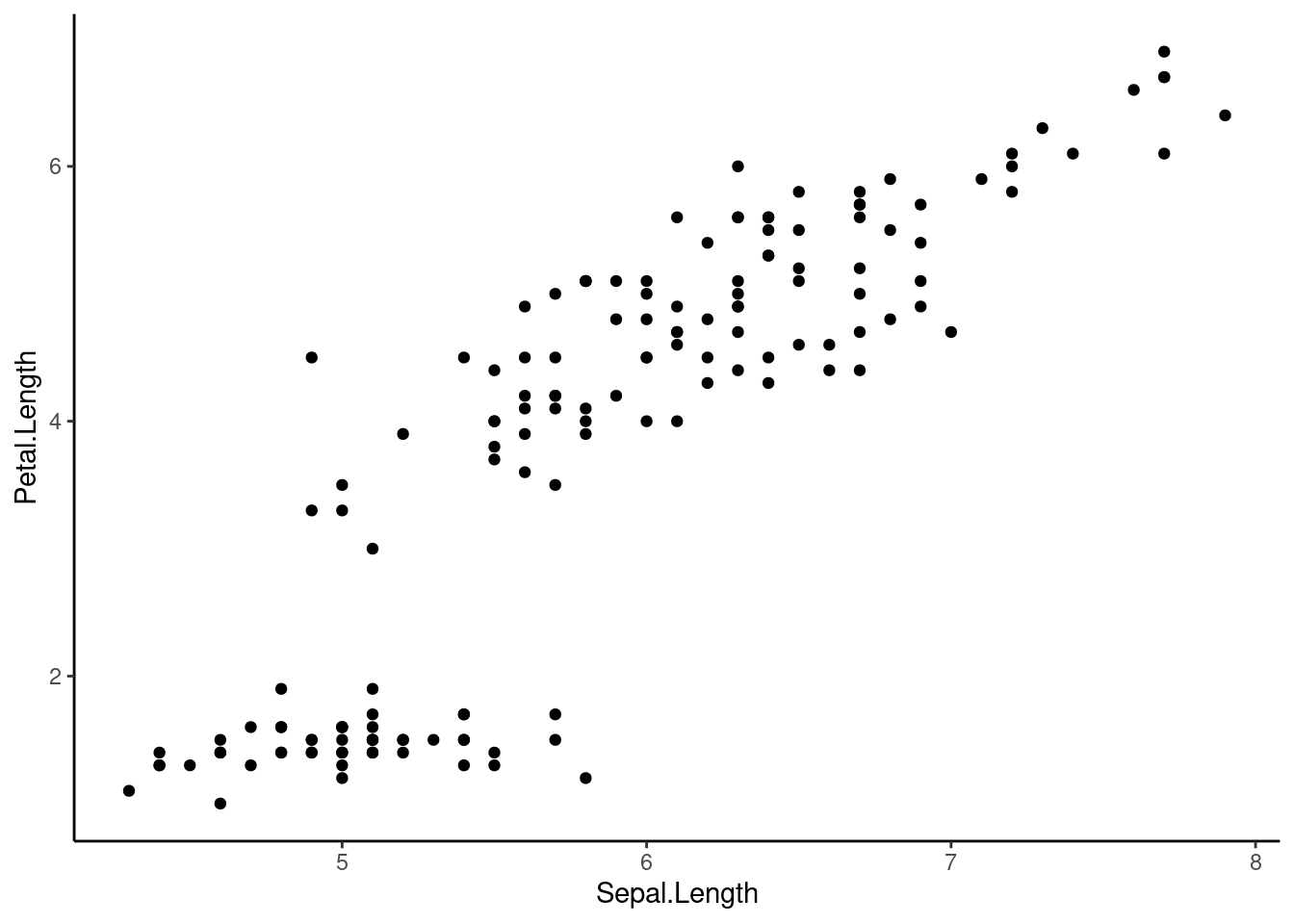
## Gráficos de dispersión
Utilicemos el conjunto de datos "iris" para crear gráficos de dispersión:
```{r}
ggplot(data = iris, mapping = aes(x = Sepal.Length, y = Petal.Length)) +
geom_point()
```
This plot is defined by 3 components: 1. "data"- iris 1. "aes" - Sepal.length vs Petal.length 1. "layer" - Points (geom)
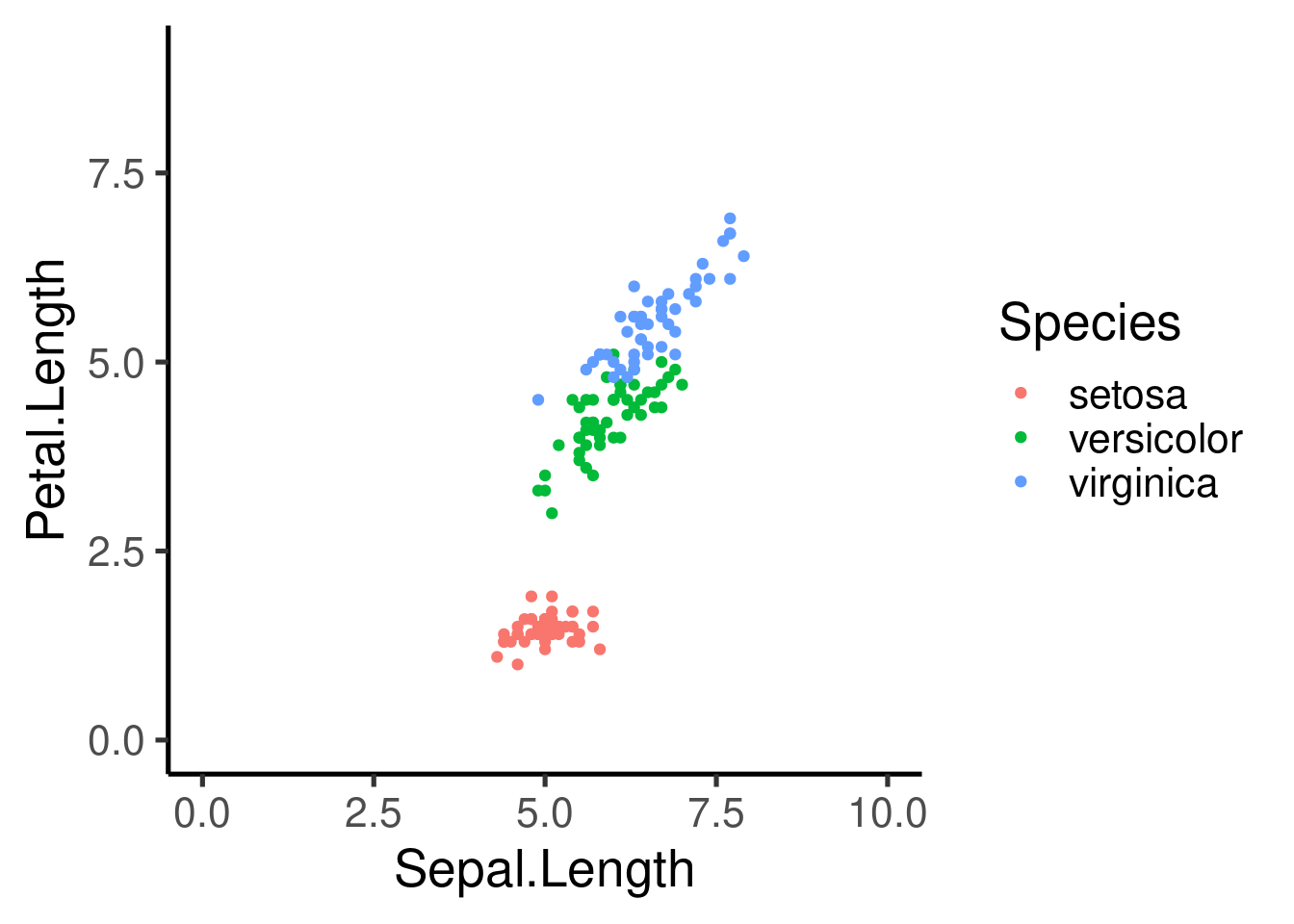
## Atributos estéticos
También podemos añadir otros atributos estéticos como el color, la forma y el tamaño. Estos atributos se pueden incluir dentro de `aes()`:
```{r}
# color by species
ggplot(
data = iris,
mapping = aes(x = Sepal.Length, y = Petal.Length, color = Species)
) +
geom_point()
# color and shape by species
ggplot(
data = iris,
mapping = aes(
x = Sepal.Length,
y = Petal.Length,
color = Species,
shape = Species
)
) +
geom_point()
```
Tenga en cuenta que los argumentos estéticos también pueden incluirse en la capa "geom":
```{r}
ggplot(
data = iris,
mapping = aes(x = Sepal.Length, y = Petal.Length)
) +
geom_point(aes(color = Species, shape = Species))
```
También podemos incluir un valor fijo:
```{r}
ggplot(
data = iris,
mapping = aes(x = Sepal.Length, y = Petal.Length)
) +
geom_point(color = "red2")
```
Algunos atributos funcionan mejor con algunos tipos de datos:
- Color y forma: variables categóricas
- Tamaño: variables continuas
</br>
::: {.alert .alert-info}
<font size="5">Ejercicio 1</font>
Utilizando el conjunto de datos "hylaeformis":
```{r, eval=F, echo=T}
# lear desde el sitio del curso
hylaeformis_data <- read.csv(
paste0("https://raw.githubusercontent.com/maRce10/", "r_avanzado_2023/master/data/hylaeformis_data.csv")
)
# or bajar manualmente y leer copia local
hylaeformis_data <- read.csv("hylaeformis_data.csv", stringsAsFactors = FALSE)
head(hylaeformis_data, 20)
```
</br>
1.1 Create a scatter plot of vs "meanfreq" (mean frequency)
1.1 Crear un gráfico de dispersión de duración ("duration") vs frecuencia promedio ("meanfreq")
```{r, eval = FALSE, echo = FALSE}
ggplot(
data = hylaeformis_data,
mapping = aes(x = duration, y = meanfreq)
) +
geom_point()
```
</br>
1.2 Añadir un atributo estético para mostrar un color diferente para cada localidad
```{r, eval = FALSE, echo = FALSE}
ggplot(
data = hylaeformis_data,
mapping = aes(x = duration, y = meanfreq)
) +
geom_point(aes(color = Locality))
```
</br>
1.3 Añade otro atributo estético para mostrar el rango de frecuencia dominante ("dfrange") como tamaño los símbolos
```{r, eval = FALSE, echo = FALSE}
ggplot(
data = hylaeformis_data,
mapping = aes(x = duration, y = meanfreq)
) +
geom_point(aes(color = Locality, size = dfrange))
```
</br>
:::
## Gráficos multipanel (Facetting)
- Otra forma de visualizar variables categóricas
- Permite crear gráficos multipanel para cada nivel de la variable
- 2 tipos: "grid" & "wrap"
```{r}
ggplot(iris, aes(Sepal.Length, Petal.Length)) +
geom_point() +
facet_wrap(~Species)
# o
ggplot(iris, aes(Sepal.Length, Petal.Length)) +
geom_point() +
facet_grid(~Species)
```
La escala puede ser fija o libre para los ejes *x* e *y*, y puede modificarse el número de columnas y filas:
```{r}
# free x
ggplot(iris, aes(Sepal.Length, Petal.Length)) +
geom_point() +
facet_wrap(~Species, scales = "free_x")
```
```{r, fig.height=8}
# free x and 3 rows
ggplot(iris, aes(Sepal.Length, Petal.Length)) +
geom_point() +
facet_wrap(~Species, scales = "free_y", nrow = 3)
```
```{r, fig.height=6}
# both free and 2 rows
ggplot(iris, aes(Sepal.Length, Petal.Length)) +
geom_point() +
facet_wrap(~Species, scales = "free", nrow = 2)
```
Tenga en cuenta que también podemos guardar el componente básico como un objeto **R** y añadir otros componentes más adelante en el código:
```{r, fig.height=8}
p <- ggplot(iris, aes(Sepal.Length, Petal.Length)) +
geom_point()
p + facet_wrap(~Species, scales = "free_x", nrow = 3)
```
## "geoms" adicionales
- `geom_smooth()` - añade las líneas de mejor ajuste (incluyendo CI)
- `geom_boxplot()` - Distribución de frecuencias
- `geom_histogram()` & `geom_freqpoly()` - distribuciones de frecuencia
- `geom_bar()` - distribución de frecuencias de variables categóricas
- `geom_path()` & `geom_line()` - añade líneas a los gráficos de dispersión
### geom_smooth()
Las líneas de regresión de mejor ajuste pueden añadirse con `geom_smooth()`:
```{r, fig.height=8}
# con CI
ggplot(iris, aes(Sepal.Length, Petal.Length)) +
geom_point() +
geom_smooth(method = "lm") +
facet_wrap(~Species, scales = "free", nrow = 3)
# sin CI
ggplot(iris, aes(Sepal.Length, Petal.Length)) +
geom_point() +
geom_smooth(method = "lm", se = FALSE) +
facet_wrap(~Species, scales = "free", nrow = 3)
```
::: {.alert .alert-info}
<font size="5">Ejercicio 2</font>
Utilizando el conjunto de datos de ejemplo "msleep":
</br>
2.1 Crear un gráfico de dispersión de peso corporal ("bodywt") frente a peso cerebral ("brainwt")
```{r, eval = FALSE, echo = FALSE}
data(msleep)
ggplot(
data = msleep,
mapping = aes(x = bodywt, y = brainwt)
) +
geom_point()
```
</br>
2.2 Añadir orden ("order") como estética del color
```{r, eval = FALSE, echo = FALSE}
ggplot(
data = msleep,
mapping = aes(x = bodywt, y = brainwt, color = order)
) +
geom_point()
```
</br>
2.3 Añadir un componente "faceta" para dividir los gráficos por orden utilizando escalas libres
```{r, eval = FALSE, echo = FALSE}
ggplot(
data = msleep,
mapping = aes(x = bodywt, y = brainwt)
) +
geom_point() +
facet_wrap(~order, scales = "free")
```
</br>
2.4 Elimine los órdenes con menos de 4 especies en el conjunto de datos y haga un gráfico similar al 2.3
```{r, eval = FALSE, echo = FALSE}
tab <- table(msleep$order)
ms2 <- msleep[msleep$order %in% names(tab)[tab >= 5], ]
ggplot(
data = ms2,
mapping = aes(x = bodywt, y = brainwt)
) +
geom_point() +
geom_smooth(method = "lm") +
facet_wrap(~order, scales = "free")
```
</br>
2.5 Añadir una línea de mejor ajuste a cada gráfico del panel
```{r, eval = FALSE, echo = FALSE}
ggplot(data = msleep[msleep$order %in% names(tab)[tab > 3], ], mapping = aes(x = bodywt, y = brainwt)) +
geom_point() +
geom_smooth() +
facet_wrap(~order, scales = "free")
```
</br>
:::
### Boxplots
De nuevo, sólo se necesita un nuevo componente "geom" para crear un boxplot:
```{r}
ggplot(iris, aes(Species, Petal.Length)) +
geom_boxplot()
```
Una alternativa interesante son los gráficos de violines:
```{r}
ggplot(iris, aes(Species, Petal.Length)) +
geom_violin()
```
### Histogramas
Lo mismo ocurre con los histrogramas y los gráficos de frecuencias:
```{r}
ggplot(iris, aes(Petal.Length)) +
geom_histogram()
ggplot(iris, aes(Petal.Length)) +
geom_freqpoly()
ggplot(iris, aes(Petal.Length)) +
geom_histogram() +
geom_freqpoly()
```
Podemos controlar la anchura de las barras:
```{r}
ggplot(iris, aes(Petal.Length)) +
geom_histogram(binwidth = 1, fill = adjustcolor("red2", alpha.f = 0.3))
```
Y compara la distribución de los distintos grupos dentro del mismo histograma:
```{r}
ggplot(iris, aes(Petal.Length, fill = Species)) +
geom_histogram(binwidth = 0.4)
```
### Gráfico de barras
Muestran la distribución de variables discretas (categóricas):
```{r}
tab <- table(msleep$order)
df <-
as.data.frame(table(msleep$order[msleep$order %in% names(tab)[tab > 3]]))
ggplot(df, aes(Var1, Freq)) +
geom_bar(stat = "identity")
```
```{r, eval=F, echo=F}
# Calcular el promedio del # de reclutas por estación
df2 <- aggregate(RECRUITS ~ SEASON, df, mean)
names(df2) <- c("season", "rec")
# Gráfico de barras
p <- ggplot(df2, aes(season, rec)) +
geom_bar(stat = "identity")
p
# Cambiar la orientación
p + coord_flip()
### Cambiar el ancho y colores de las barras ###
# Cambiar el ancho de las barras
ggplot(df2, aes(season, rec)) +
geom_bar(stat = "identity", width = 0.5)
ggplot(df2, aes(season, rec)) +
geom_bar(stat = "identity", width = 0.2)
# Cambiar colores
ggplot(df2, aes(season, rec)) +
geom_bar(stat = "identity", color = "blue", fill = "white")
# Remover el tema y agregar barras azules
p <- ggplot(df2, aes(season, rec)) +
geom_bar(stat = "identity", fill = "steelblue") +
theme_minimal()
p
# Escoger que barras mostrar
p + scale_x_discrete(limits = c("Spring", "Winter"))
### Agregar anotaciones ###
df2$rec <- round(df2$rec, 1)
# Fuera de las barras
ggplot(df2, aes(season, rec)) +
geom_bar(stat = "identity", fill = "steelblue") +
geom_text(aes(label = rec), vjust = -0.3, size = 3.5) +
theme_minimal()
# Dentro de las barras
ggplot(df2, aes(season, rec)) +
geom_bar(stat = "identity", fill = "yellow") +
geom_text(aes(label = rec), vjust = 1.6, size = 3.5) +
theme_minimal()
### Cambiar colores de barras por grupo ###
# Cambiar el color de las líneas externas por grupo
p <- ggplot(df2, aes(season, rec, color = season)) +
geom_bar(stat = "identity", fill = "white")
p
### Cambiar el color manualmente ###
# Usar "palette" predefinido de colores
p + scale_color_manual(values = c("red", "blue", "green", "yellow"))
# Usar "palette brewer"
p + scale_color_brewer(palette = "Dark2")
p + scale_color_brewer(palette = 1)
p + scale_color_brewer(palette = 2)
# Usar escala de grises
p + scale_color_grey() + theme_classic()
### Cambiar colores de relleno de las barras ###
# Color de relleno
p <- ggplot(df2, aes(season, rec, fill = season)) +
geom_bar(stat = "identity") +
theme_minimal()
p
### Cambiar el color de relleno manualmente ###
# Usar "palette" predefinido de colores
p + scale_fill_manual(values = c("red", "blue", "green", "yellow"))
# Usar "palette brewer"
p + scale_fill_brewer(palette = "Dark2")
p + scale_fill_brewer(palette = 1)
p + scale_fill_brewer(palette = 2)
# Usar escala de grises
p + scale_fill_grey() + theme_classic()
# Usar color negro del borde y relleno
ggplot(df2, aes(season, rec, fill = season)) +
geom_bar(stat = "identity", color = "black") +
scale_fill_manual(values = c("red", "blue", "green", "yellow")) +
theme_classic()
### Cambiar la posición de la leyenda ###
# Cambiar el color de las barras a tonos de azúl
p <- ggplot(df2, aes(season, rec, fill = season)) +
geom_bar(stat = "identity") +
theme_minimal()
p1 <- p + scale_fill_brewer(palette = "Blues")
p1 + theme(legend.position = "top")
p1 + theme(legend.position = "bottom")
# Remover la leyenda
p1 + theme(legend.position = "none")
### Cambiar el órden de los items en una leyenda ###
# Cambiar órden de grupos
p1 + scale_x_discrete(limits = c("Summer", "Autumn", "Winter", "Spring"))
### Editar aún más los elementos de la leyenda ###
# Cambiar el título de la leyenda y la fuente
p1 <- ggplot(df2, aes(season, rec, fill = season)) +
geom_bar(stat = "identity", color = "black")
titulo <- "Estación"
p2 <- p1 + scale_fill_manual(titulo, values = c("red", "blue", "green", "yellow")) +
theme_classic()
p2 + theme(legend.title = element_text(
colour = "blue", size = 10,
face = "bold"
))
p2 + theme(legend.text = element_text(
colour = "blue", size = 10,
face = "bold"
))
# Cambiar el color del fondo de la leyenda
p2 + theme(legend.background = element_rect(
fill = "lightblue",
size = 0.5, linetype = "solid"
))
p2 + theme(legend.background = element_rect(
fill = "lightblue",
size = 0.5, linetype = "solid"
))
### Gráfico de barras con múltiples grupos ###
head(df)
df.n <- aggregate(RECRUITS ~ SEASON + DENSITY, df, mean)
names(df.n) <- c("season", "density", "rec")
# Gráfico 1
ggplot(df.n, aes(season, rec, fill = density)) +
geom_bar(stat = "identity")
# Gráfico 2
ggplot(df.n, aes(season, rec, fill = density)) +
geom_bar(stat = "identity", position = position_dodge())
# Cambiar el color manualmente
p <- ggplot(df.n, aes(season, rec, fill = density)) +
geom_bar(stat = "identity", color = "black", position = position_dodge()) +
theme_minimal()
p + theme_classic()
# Colores manuales
p + scale_fill_manual(values = c("#999999", "#E69F00")) + theme_classic()
# Palette de colores
p + scale_fill_brewer(palette = "Blues") + theme_classic()
### Gráficos de barras con error ###
# Calcular promedio +/- SD
head(df)
df3 <- aggregate(RECRUITS ~ SEASON + DENSITY, df, FUN = function(x) c(mean = mean(x), sd = sd(x)))
df4 <- cbind(data.frame(df3[, c(1:2)]), df3$RECRUITS[, 1], df3$RECRUITS[, 2])
names(df4) <- c("season", "density", "mean", "sd")
str(df4)
# Grafico de barras y desviación estándar
p <- ggplot(df4, aes(season, mean, fill = density)) +
geom_bar(stat = "identity", position = position_dodge(), color = "black") +
geom_errorbar(aes(ymin = mean, ymax = mean + sd),
width = 0.2,
position = position_dodge(0.9)
)
p + scale_fill_brewer(palette = "Paired") + theme_minimal()
p + scale_fill_brewer(palette = "Paired") + theme_classic()
### Personalizar aún más los gráficos ###
# Poner títulos de ejes
p + xlab("Estación") +
ylab("Número de reclutas") +
theme_classic()
# Remover títulos de ejes
p + xlab(NULL) +
ylab(NULL) +
theme_classic()
# Otros cambios
p2 <- p + labs(x = "Estación", y = "Número de reclutas") +
scale_fill_manual(values = c("black", "grey50")) +
theme_classic()
p2
# Cambiar los límites X y Y
p2 + ylim(0, 100)
# Cambiar límites con expand_limits
p2 + expand_limits(x = c(0, 6), y = c(0, 80))
```
## Personalización de ggplots
Además de las funciones básicas (por ejemplo, componentes) descritas anteriormente, ggplot tiene muchas otras herramientas (tanto argumentos como funciones adicionales) para personalizar aún más los gráficos. Prácticamente todo se puede modificar. Aquí vemos algunas de las herramientas más comunes.
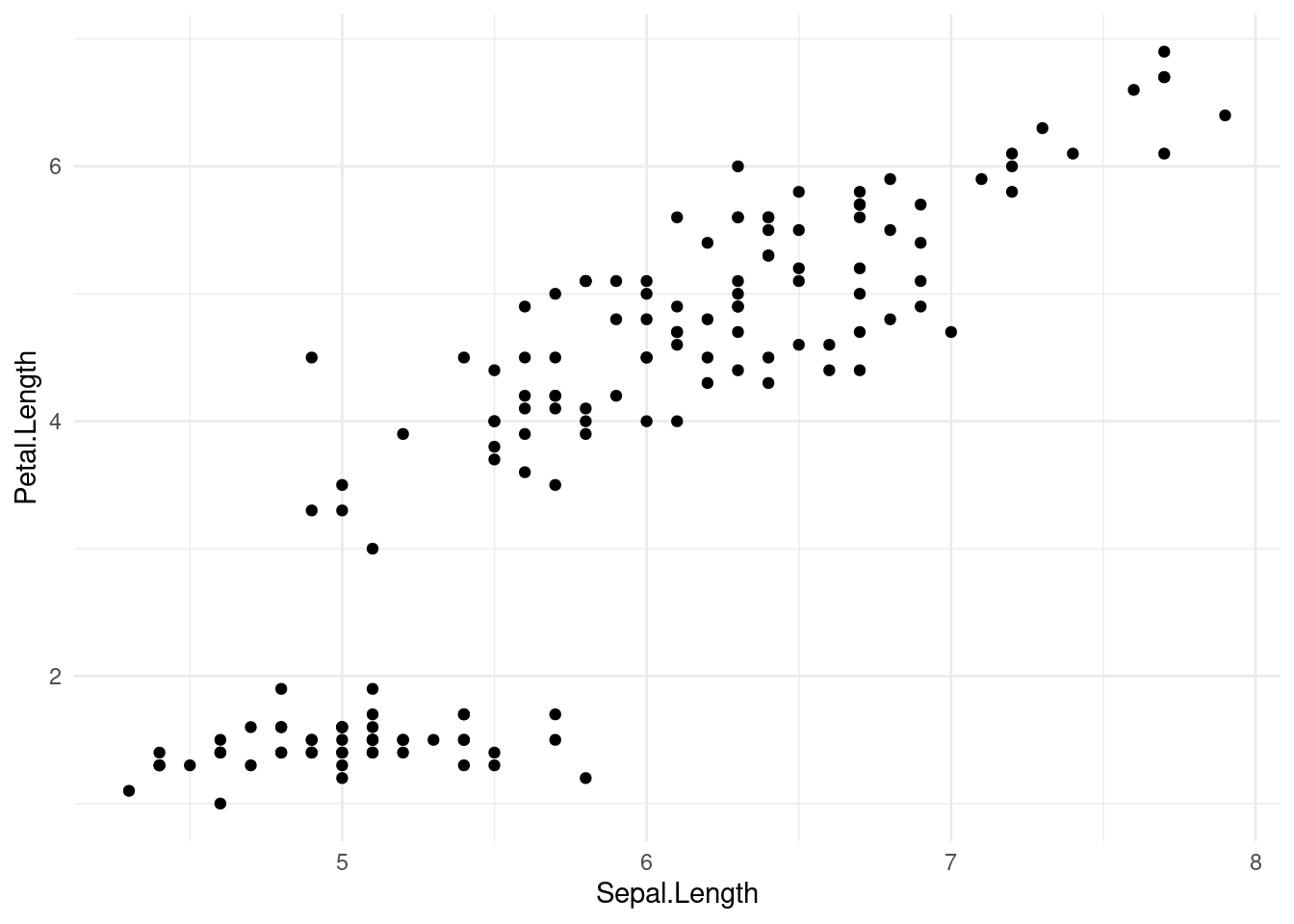
### Temas
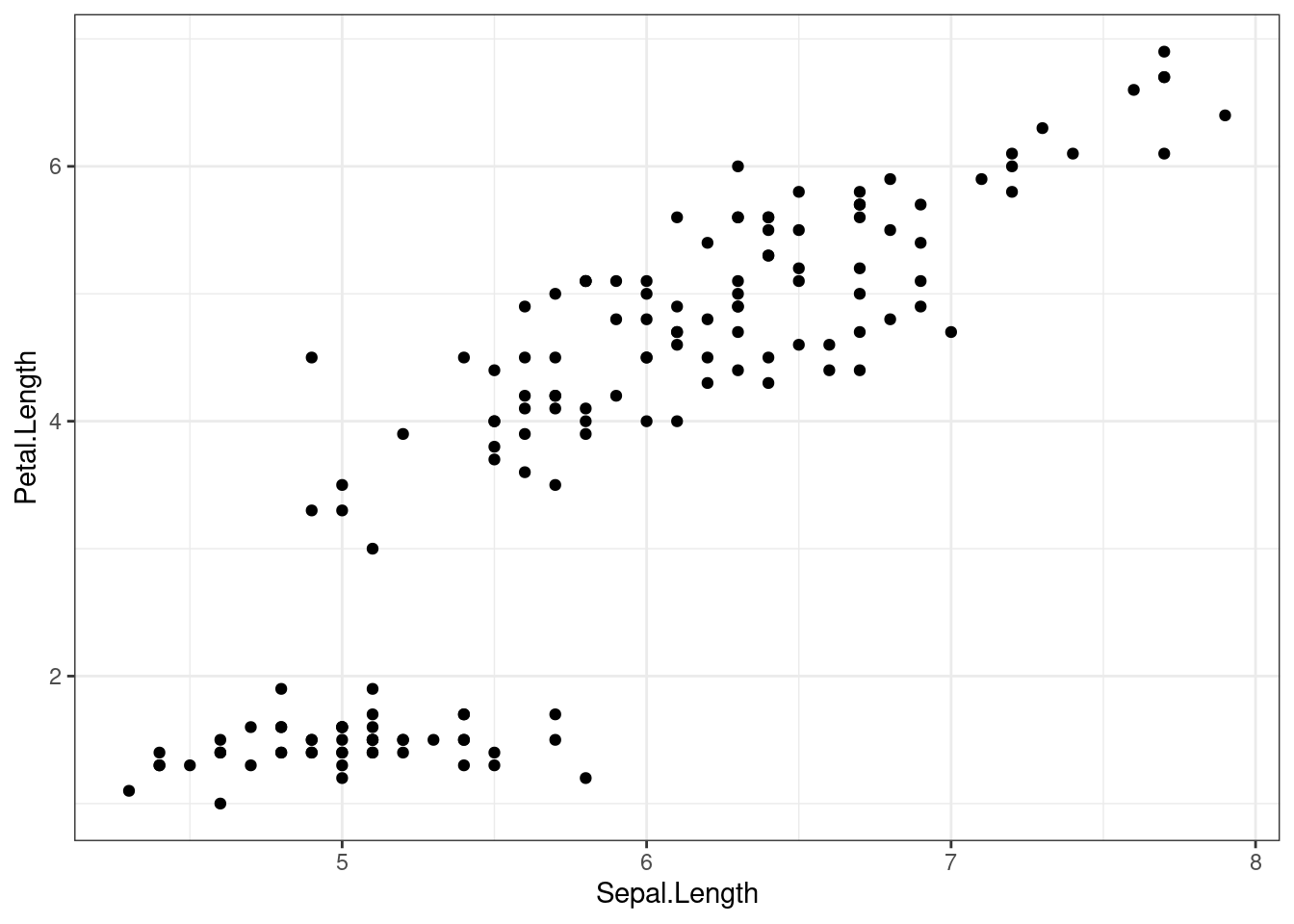
ggplot2 viene con algunos temas por defecto que se pueden aplicar fácilmente para modificar el aspecto de nuestros gráficos:
```{r}
p <- ggplot(iris, aes(Sepal.Length, Petal.Length)) +
geom_point()
p + theme_classic()
p + theme_bw()
p + theme_minimal()
```
La mayoría de los temas difieren en el uso de cuadrículas, líneas de borde y patrones de etiquetado de ejes.
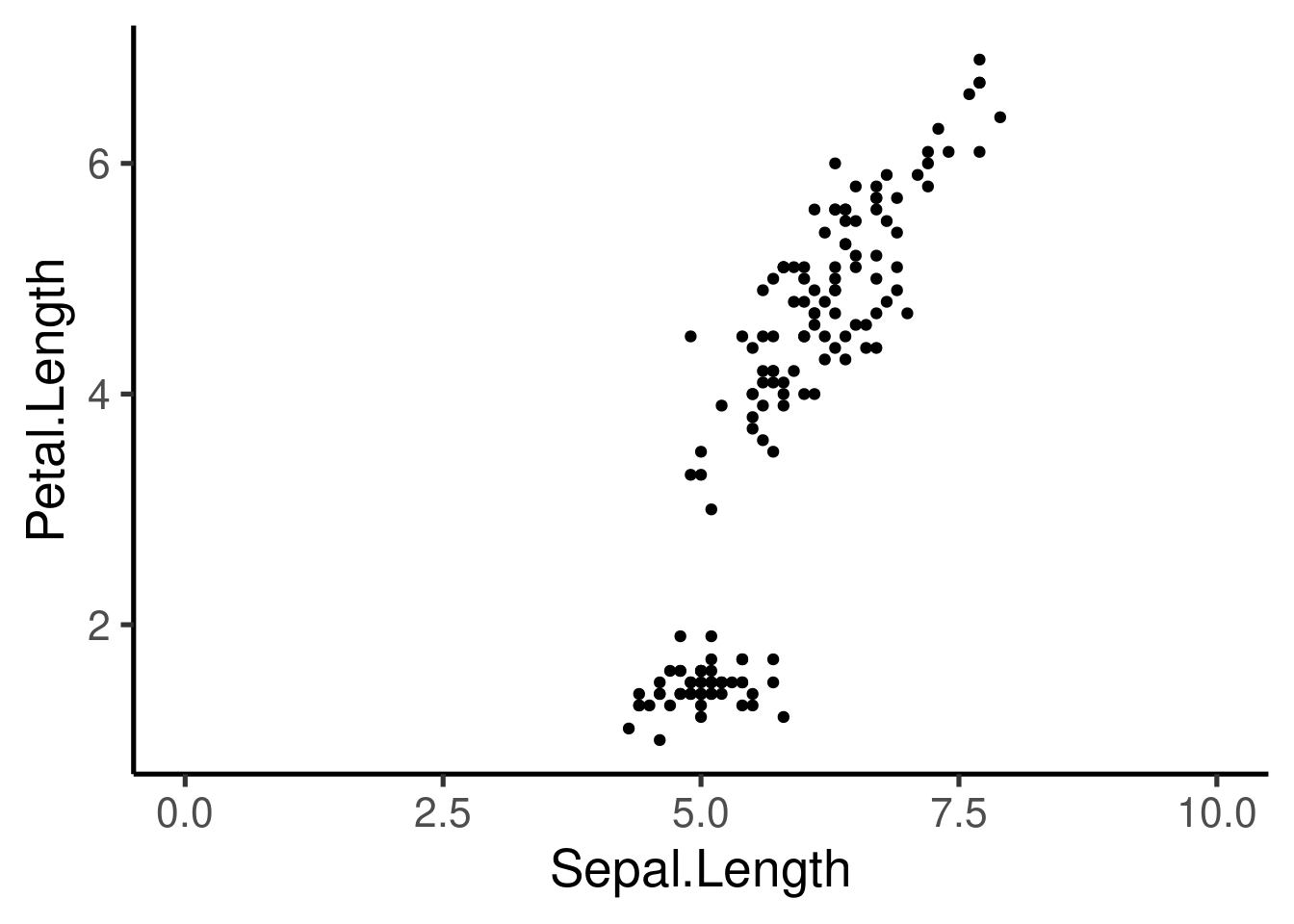
### Personalización de ejes
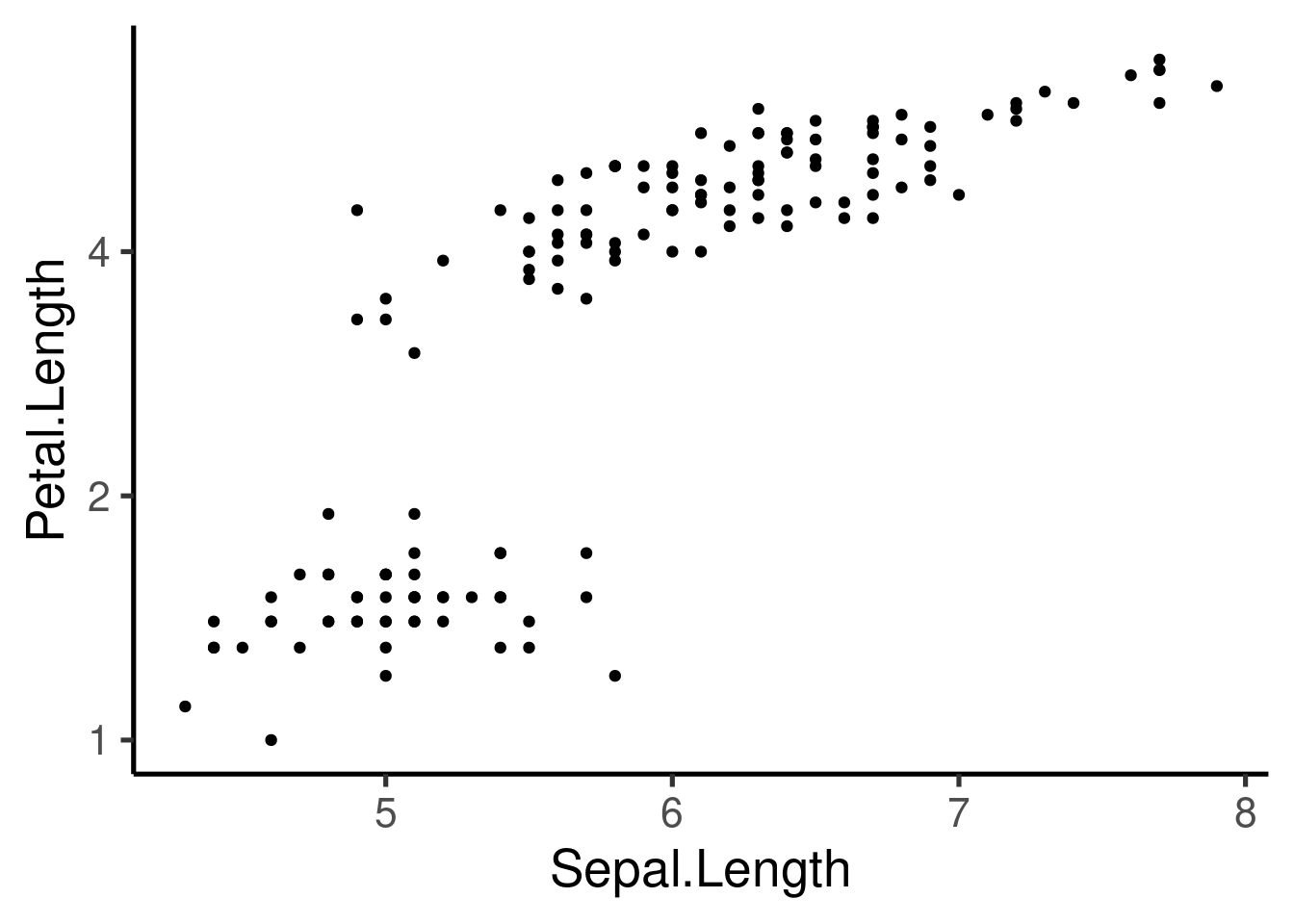
Los límites de los ejes pueden modificarse como sigue:
```{r}
ggplot(iris, aes(Sepal.Length, Petal.Length)) +
geom_point() +
xlim(c(0, 10))
ggplot(iris, aes(Sepal.Length, Petal.Length, col = Species)) +
geom_point() +
xlim(c(0, 10)) +
ylim(c(0, 9))
```
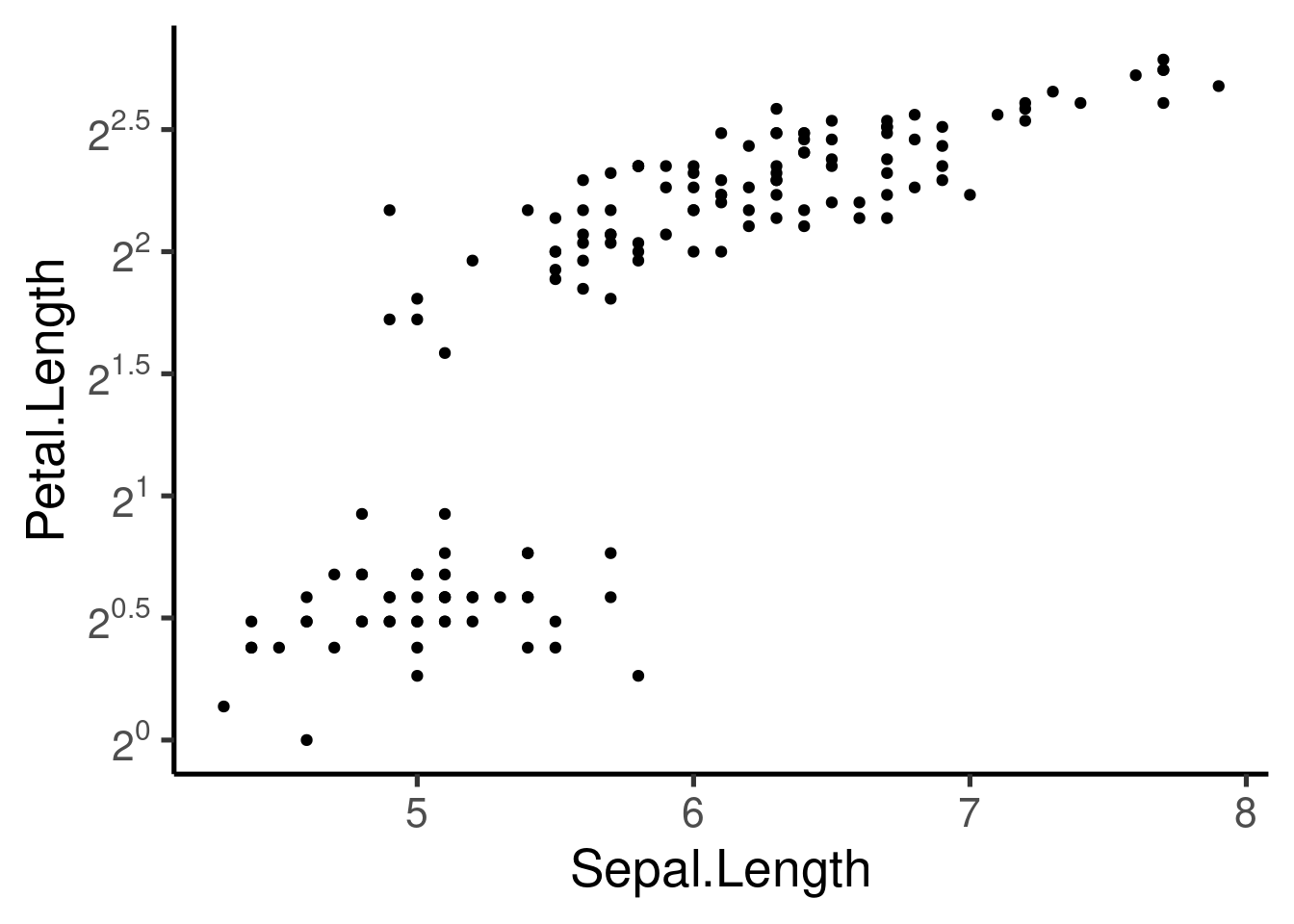
Los ejes también pueden transformarse:
```{r}
ggplot(iris, aes(Sepal.Length, Petal.Length, col = Species)) +
geom_point() +
scale_x_continuous(trans = "log") +
scale_y_continuous(trans = "log2")
```
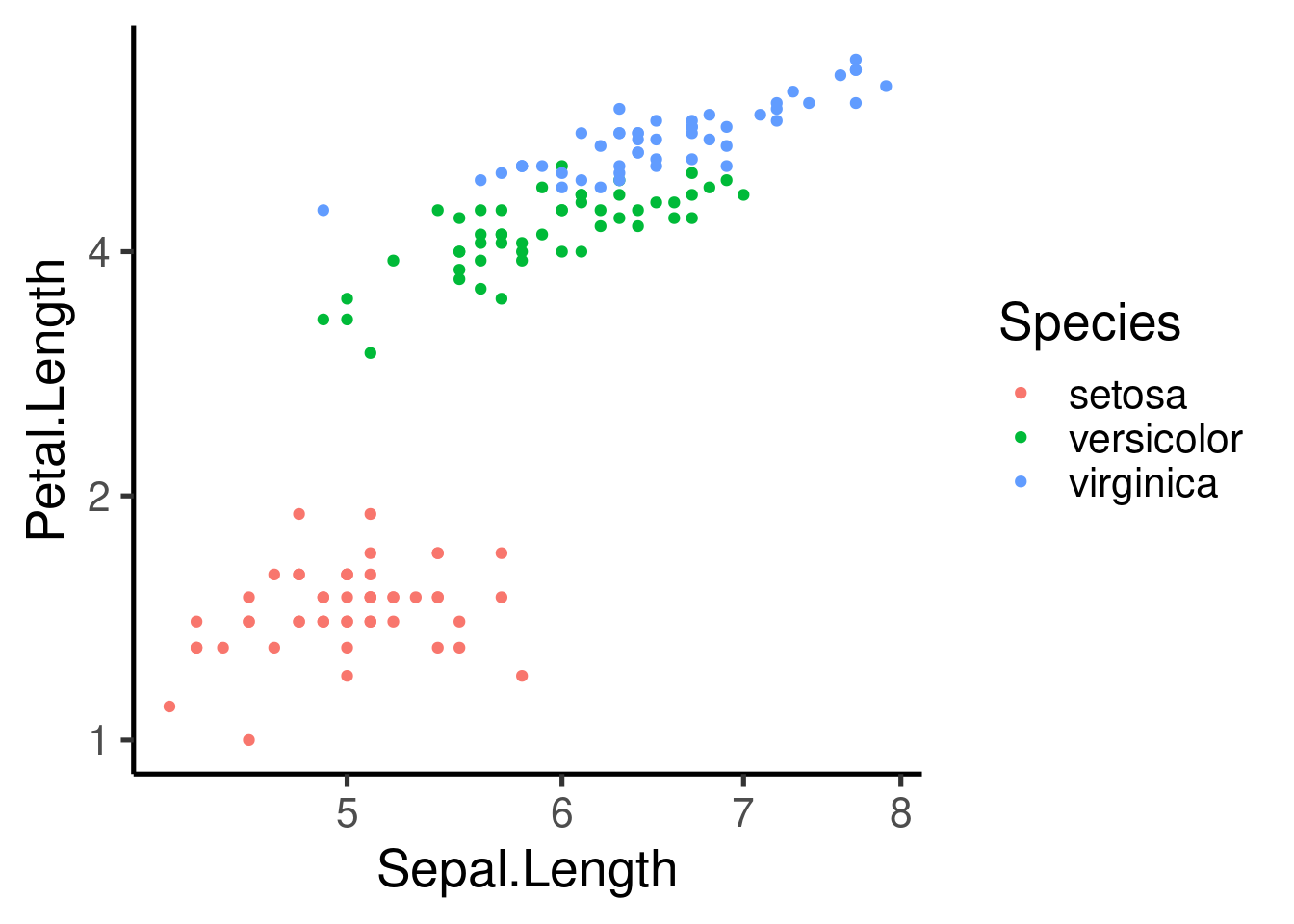
o invertidos:
```{r}
ggplot(iris, aes(Sepal.Length, Petal.Length, col = Species)) +
geom_point() +
scale_y_reverse()
```
### Guardar ggplots
Los ggplots pueden exportarse como archivos de imagen utilizando la función `ggsave`:
```{r}
ggplot(
data = msleep[msleep$order %in% names(tab)[tab > 5], ],
mapping = aes(x = bodywt, y = brainwt)
) +
geom_point() +
facet_wrap(~order, scales = "free")
# Export
ggsave("plot.png", width = 5, height = 5)
```
El tipo de archivo de imagen se identificará por la extensión en el nombre del archivo
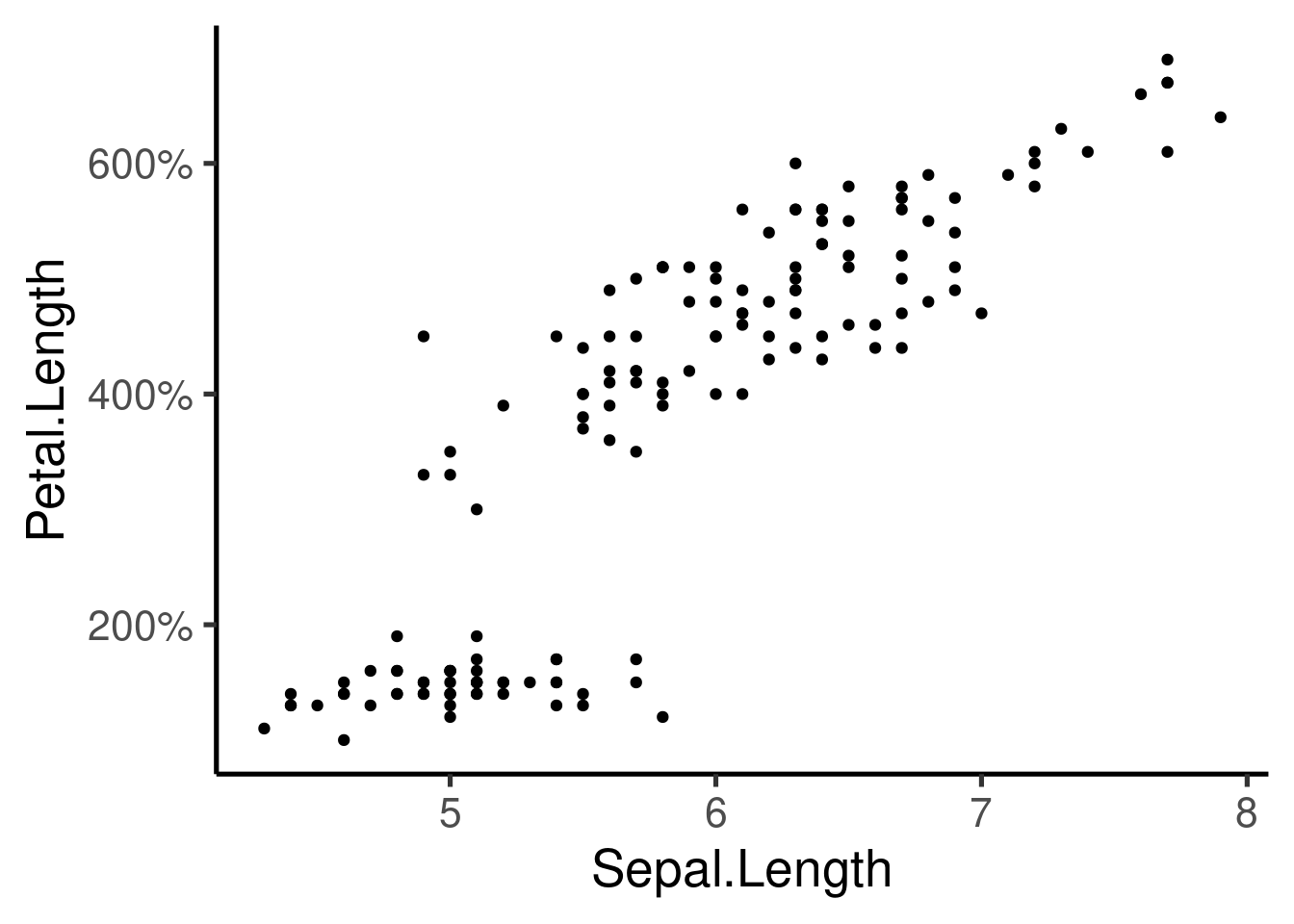
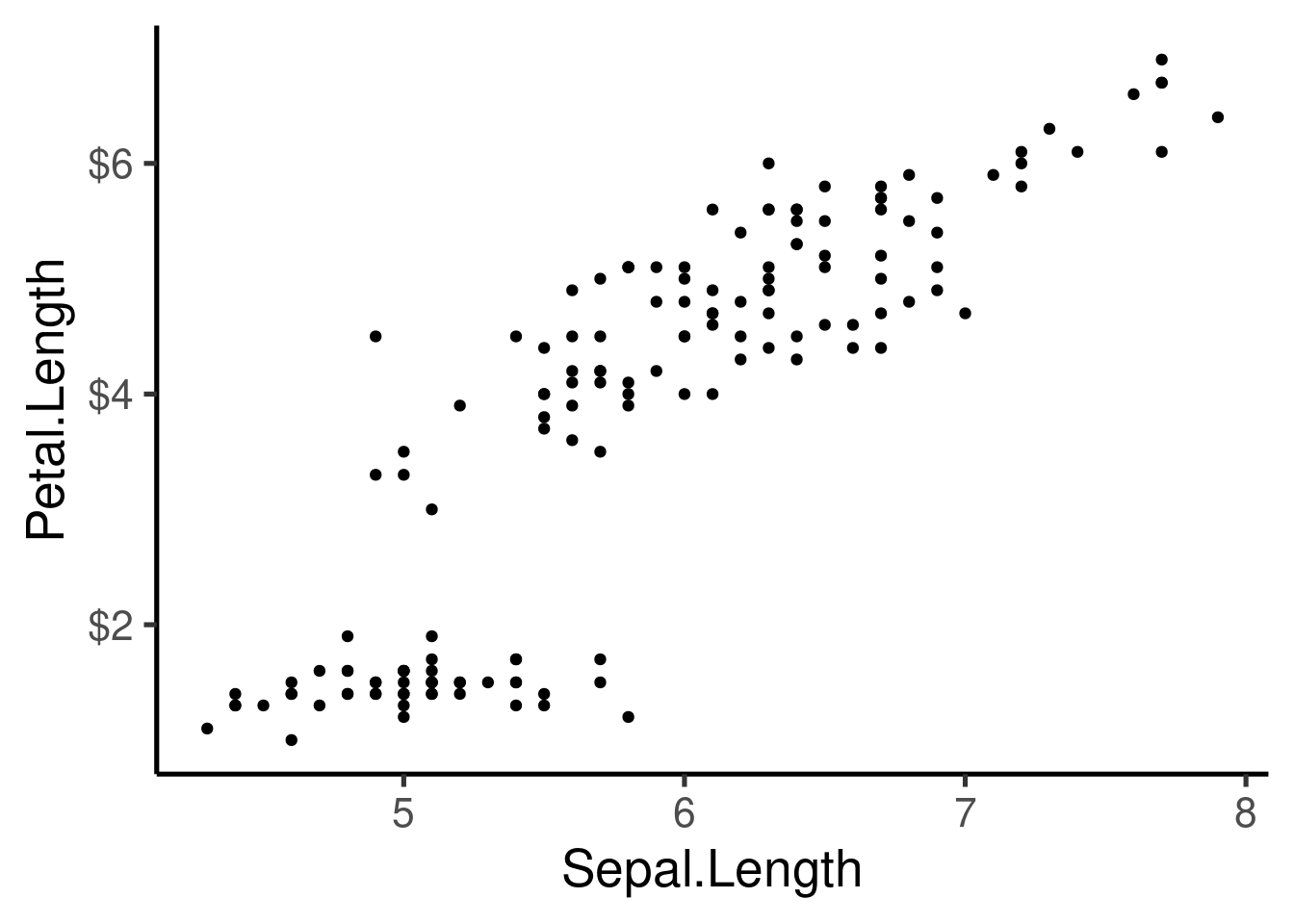
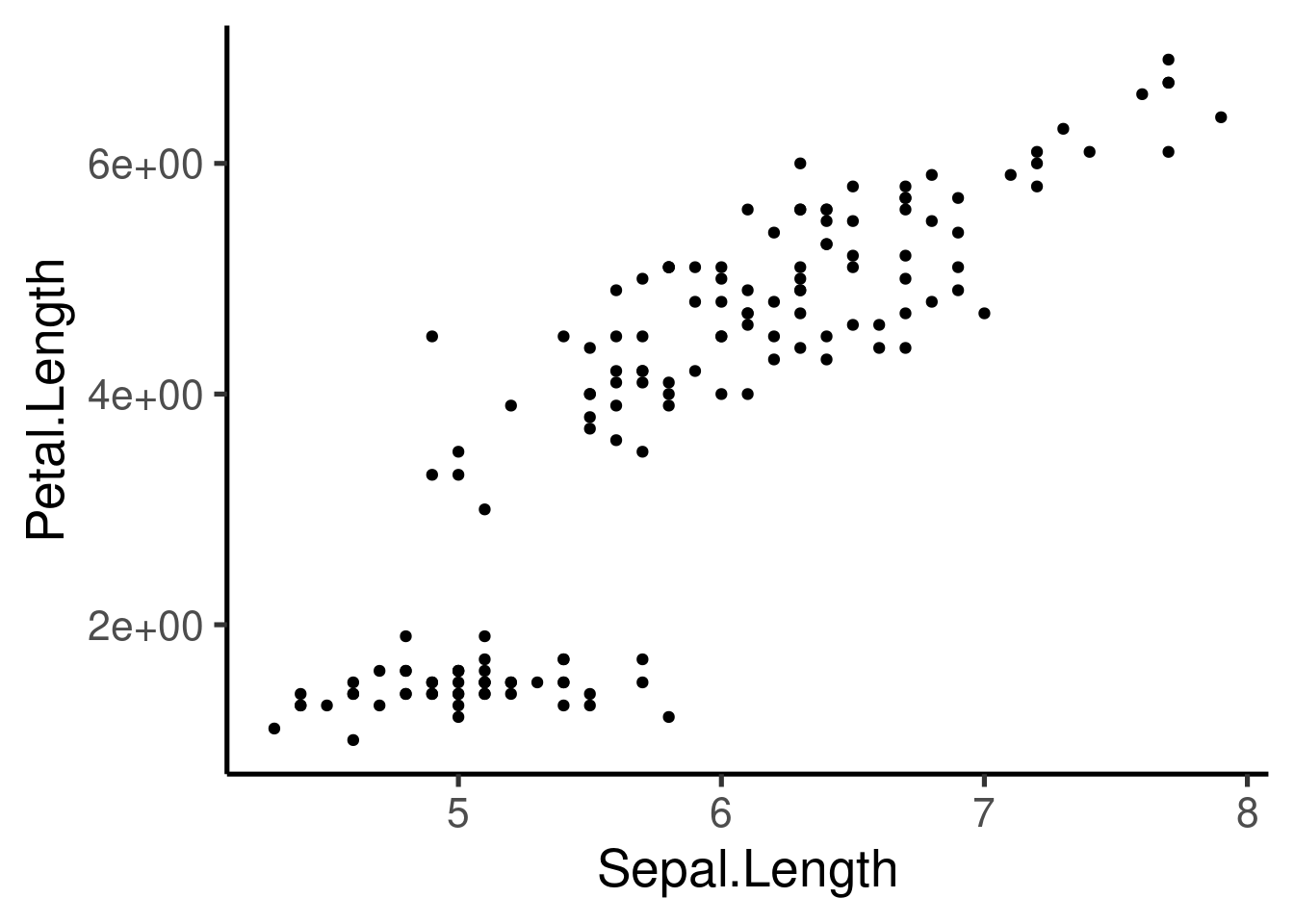
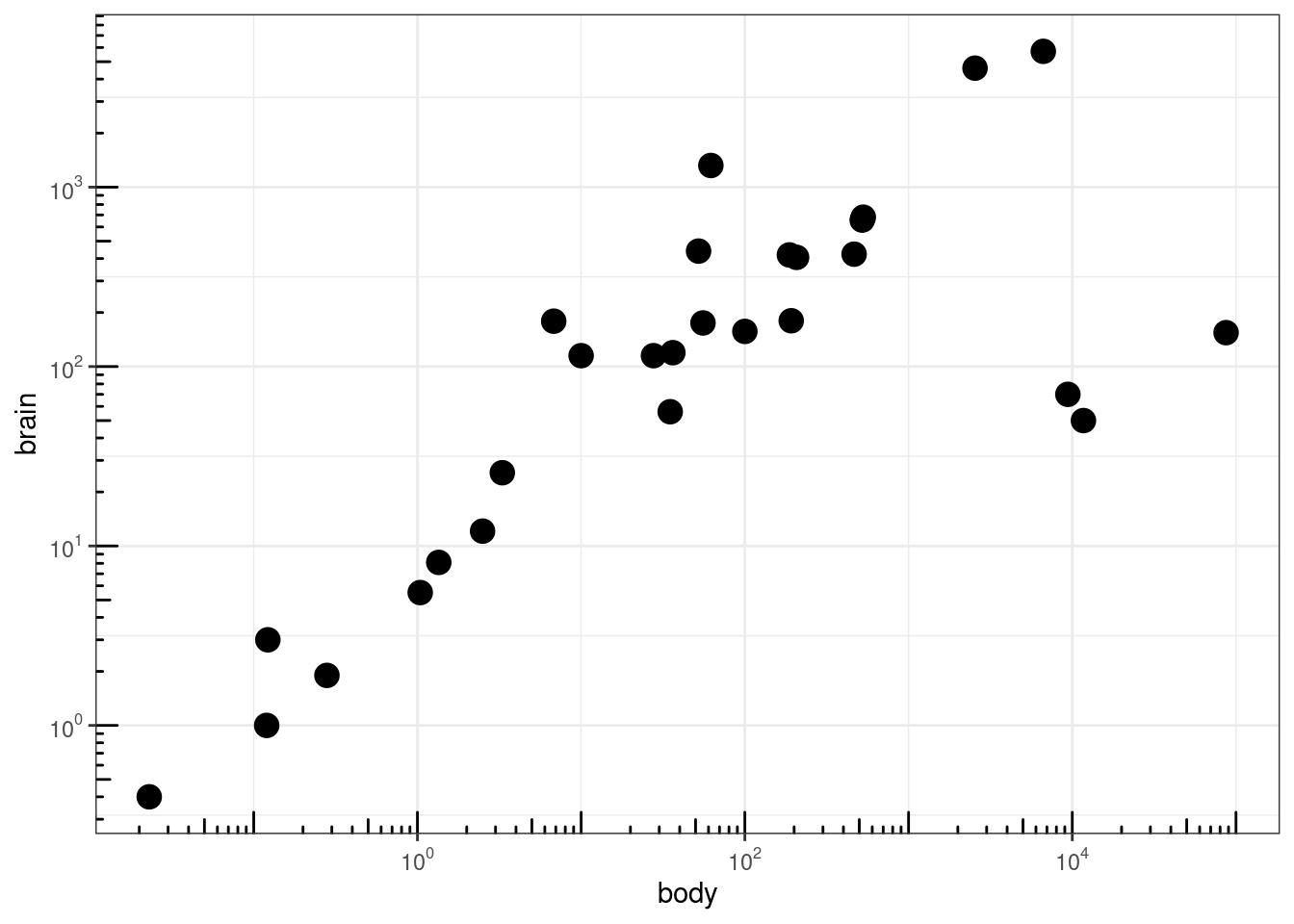
Personalización adicional del eje:
```{r}
# Log2 scaling of the y axis (with visually-equal spacing)
require(scales)
p + scale_y_continuous(trans = log2_trans())
# show exponents
p + scale_y_continuous(
trans = log2_trans(),
breaks = trans_breaks("log2", function(x) 2^x),
labels = trans_format("log2", math_format(2^.x))
)
# Percent
p + scale_y_continuous(labels = percent)
# dollar
p + scale_y_continuous(labels = dollar)
# scientific
p + scale_y_continuous(labels = scientific)
### Agregar "tick marks" ###
# Cargar librerías
library(MASS)
data(Animals)
# x and y axis are transformed and formatted
p2 <- ggplot(Animals, aes(x = body, y = brain)) +
geom_point(size = 4) +
scale_x_log10(
breaks = trans_breaks("log10", function(x) 10^x),
labels = trans_format("log10", math_format(10^.x))
) +
scale_y_log10(
breaks = trans_breaks("log10", function(x) 10^x),
labels = trans_format("log10", math_format(10^.x))
) +
theme_bw()
# log-log plot without log tick marks
p2
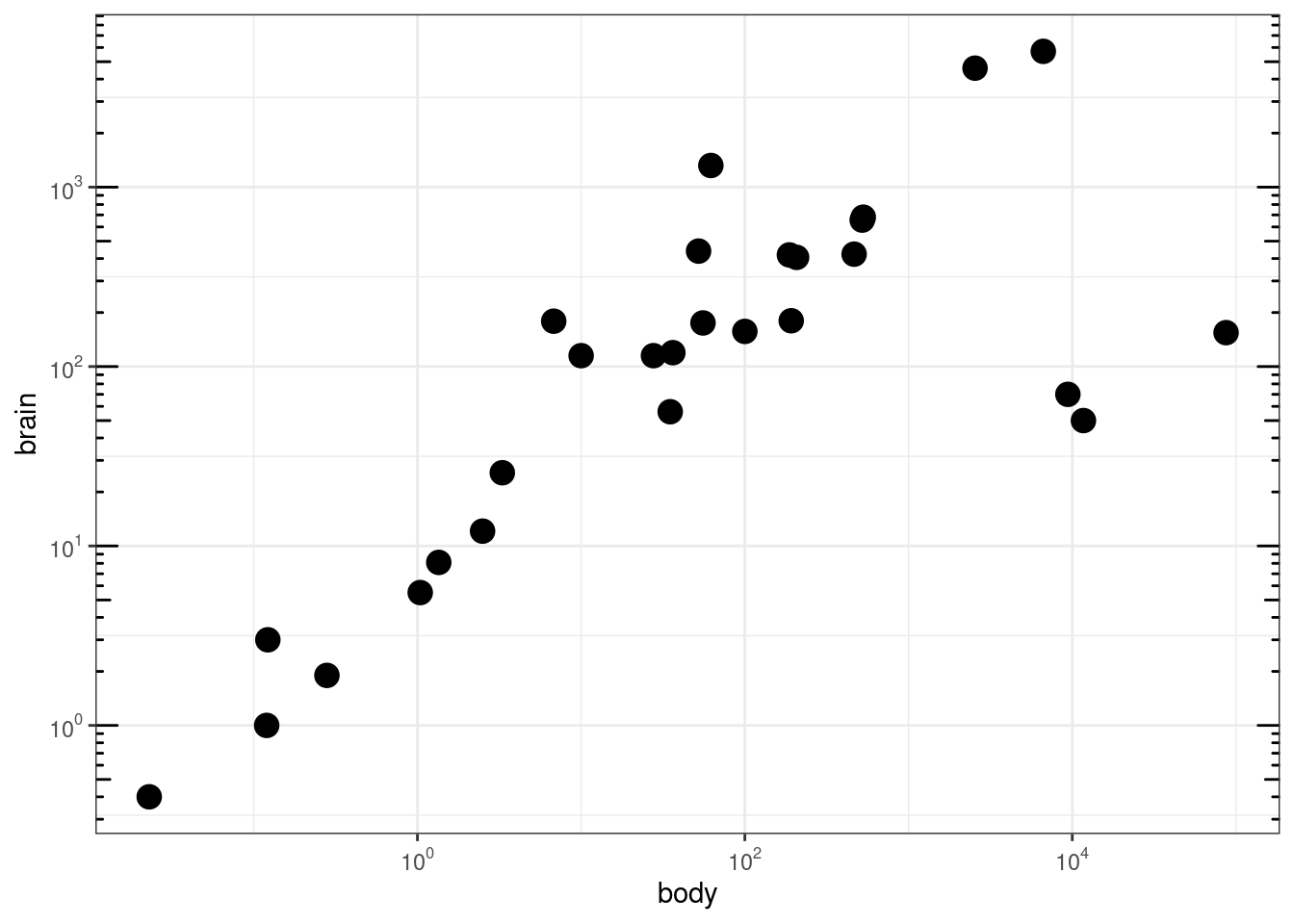
# Show log tick marks
p2 + annotation_logticks()
# # Log ticks on left and right
p2 + annotation_logticks(sides = "lr")
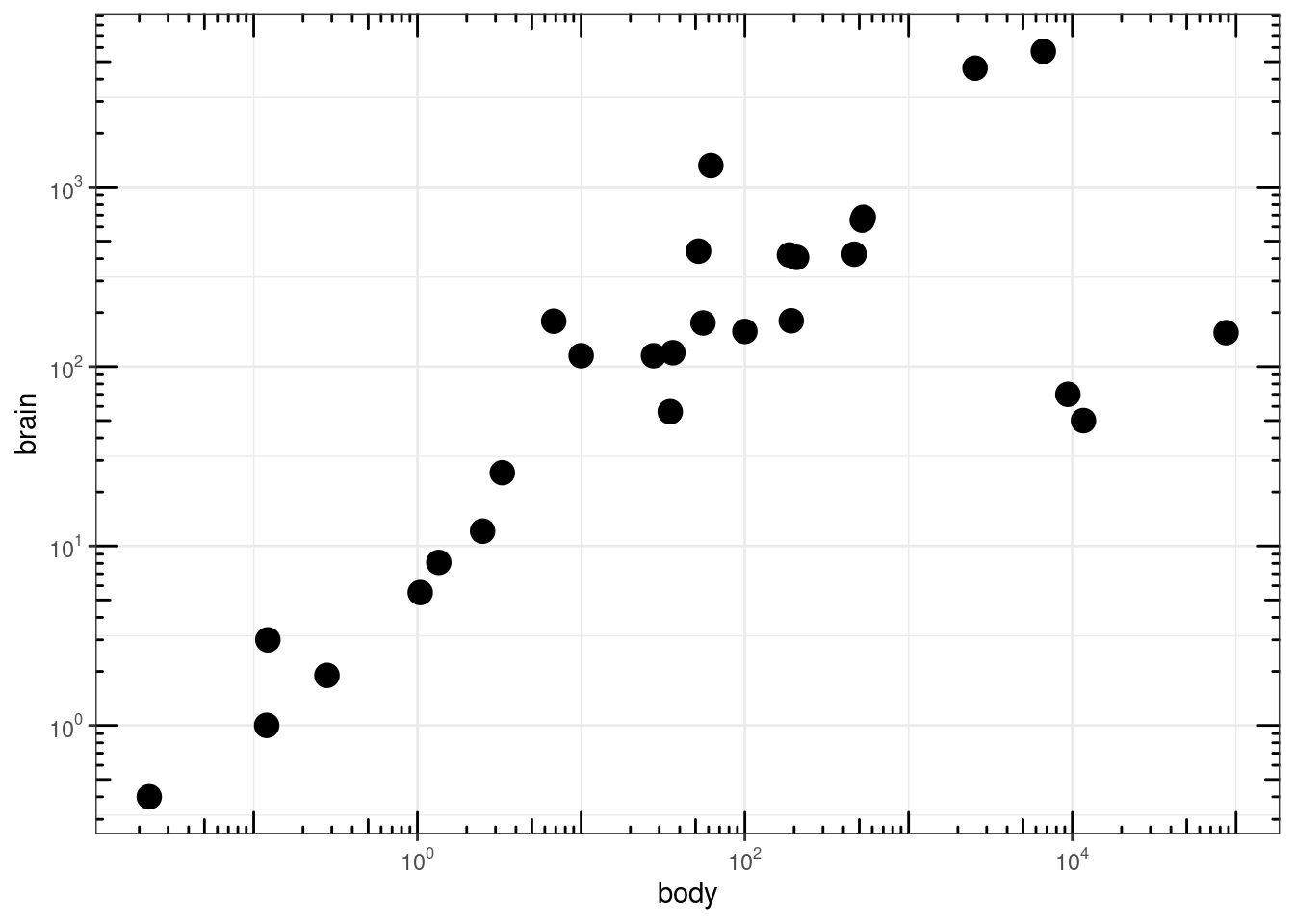
# All sides
p2 + annotation_logticks(sides = "trbl")
```
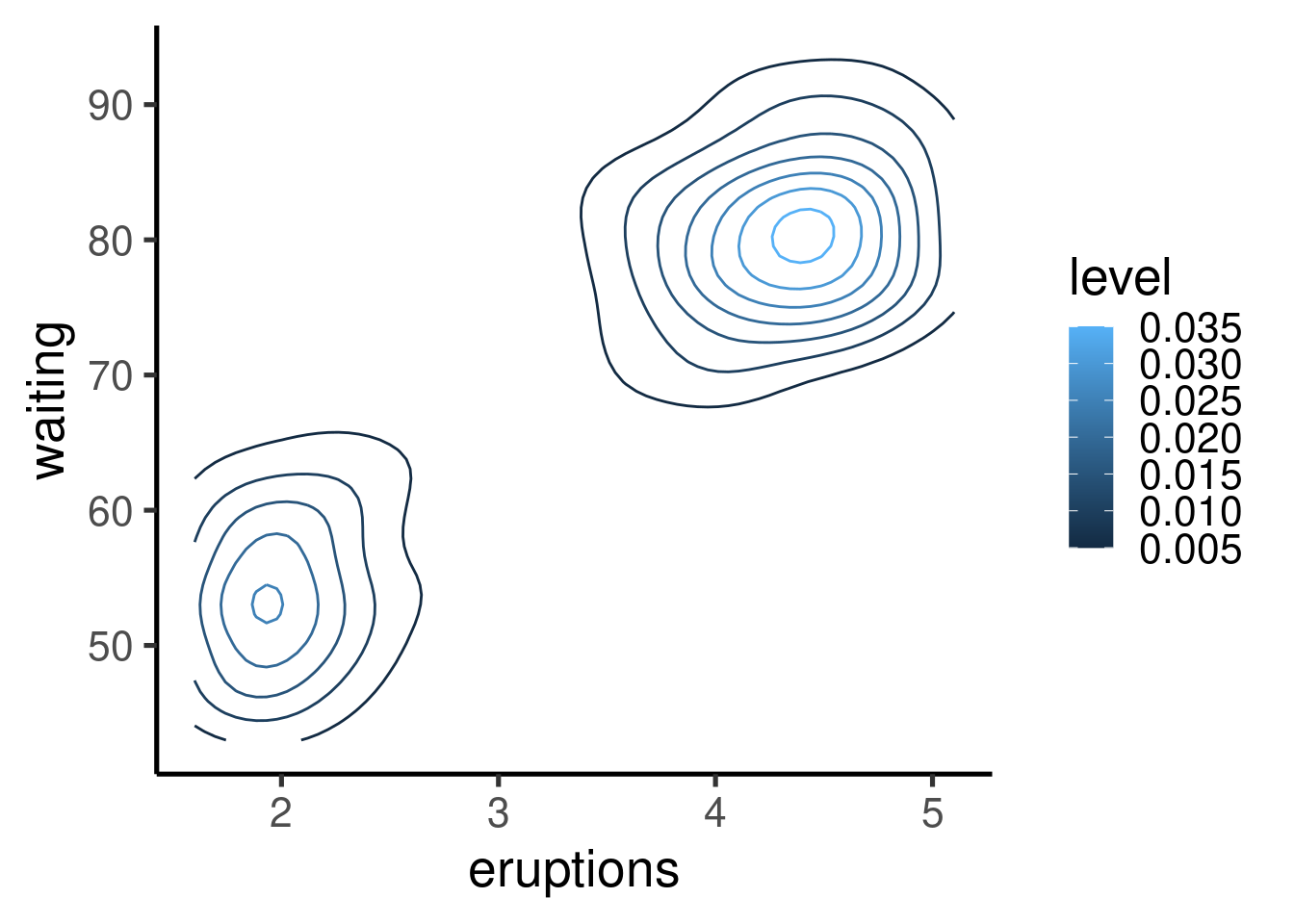
### Otros gráficos
Se pueden generar muchos otros tipos de gráficos. Aquí muestro un ejemplo de gráficos de contorno y de "mapa de calor":
```{r}
head(faithful)
ggplot(faithfuld, aes(eruptions, waiting)) +
geom_contour(aes(z = density, colour = after_stat(level)))
ggplot(faithfuld, aes(eruptions, waiting)) +
geom_raster(aes(fill = density))
```
## Otros paquetes de gráficos en R
- **ggvis** (ggplots interactivos)
- **vcd** (Warnes 2015)
- **plotrix** (Lemon et al. 2006)
- **gplots** (Warnes 2015)
Consulte la [CRAN Graphics Task View](http://cran.r-project.org/web/views/Graphics.html) para obtener una lista más completa de herramientas gráficas en **R**.
------------------------------------------------------------------------
## References
- Lemon J (2006) Plotrix: a package in the red light district of R. R-News 6(4):8–12
- Warnes GR, Bolker B, Bonebakker L, Gentleman R, Liaw WHA, Lumley T, Maechler M, Magnusson A, Moeller S, Schwartz M, Venables B (2015) gplots: various R programming tools for plotting data. R package version 2.17.0. https://CRAN.R-project.org/package=gplots
- Wickham H (2010) A layered grammar of graphics. J Comput Graph Stat 19(1):3–28
- Wilkinson L (2005) The grammar of graphics. Statistics and computing, 2nd edn. Springer, New York
------------------------------------------------------------------------
------------------------------------------------------------------------
<font size="4">Información de la sesión</font>
```{r session info, echo=F}
sessionInfo()
```